La celulosa es la fuente de carbono renovable más abundante de la Tierra. Sin embargo, la estructura de este polímero constituye una barrera física y química para acceder al carbono, lo que ha limitado el aprovechamiento del mismo. En la naturaleza, un pequeño porcentaje de microorganismos pueden degradarla a través de la expresión de celulasas. Dentro de estos microorganismos, uno de los grupos más activos y eficientes son los hongos filamentosos. Esta revisión describe las similitudes y diferencias de los mecanismos de acción de las celulasas y los mecanismos de regulación de su expresión para 3 de los modelos de hongos filamentosos celulolíticos más estudiados: Trichoderma reesei, Aspergillus niger y Aspergillus nidulans, y para un modelo recientemente descrito, Neurospora crassa. Se encontró que los mecanismos de acción enzimática son muy similares en todos los modelos estudiados, no así los mecanismos de regulación génica. Entender las particularidades de cada sistema es fundamental en el desarrollo de estrategias para la mejora de la producción de celulasas, ya sea proporcionando el ambiente óptimo (condiciones de fermentación) o aumentando la expresión en estos microorganismos mediante ingeniería genética.
Cellulose is the most abundant renewable carbon source on earth. However, this polymer structure comprises a physical and chemical barrier for carbon access, which has limited its exploitation. In nature, only a few percentage of microorganisms may degrade this polymer by cellulase expression. Filamentous fungi are one of the most active and efficient groups among these microorganisms. This review describes similarities and differences between cellulase activity mechanisms and regulatory mechanisms controlling gene expression for 3 of the most studied cellulolytic filamentous fungi models: Trichoderma reesei, Aspergillus niger and Aspergillus nidulans, and the recently described model Neurospora crassa. Unlike gene expression mechanisms, it was found that enzymatic activity mechanisms are similar for all the studied models. Understanding the distinctive elements of each system is essential for the development of strategies for the improvement of cellulase production, either by providing the optimum environment (fermentation conditions) or increasing gene expression in these microorganisms by genetic engineering.
La celulosa es la fuente de carbono renovable más abundante de la Tierra. Es sintetizada en su mayoría por las plantas a través de la fotosíntesis, con una tasa de producción anual de 7,2×1011 toneladas31. Sin embargo, este carbono no está disponible fácilmente. Se ha estimado que a pH neutro y en ausencia de enzimas la vida media de la celulosa es de millones de años72, indicando que la hidrólisis enzimática es clave en la degradación de este polímero. Aunque se ha reportado que algunos insectos, moluscos, nematodos y protozoos pueden producir celulasas, los microorganismos son los mayores productores de estas enzimas72. Se ha encontrado actividad celulolítica en una gran variedad de bacterias y hongos. Sin embargo, no todos son capaces de solubilizar la celulosa completamente5. Los hongos son reconocidos como agentes de descomposición de la materia orgánica en general y de la celulosa en particular, para lo cual producen una gran variedad de enzimas hidrolíticas esenciales para soportar su crecimiento, ya sea como saprófitos o como patógenos2. La actividad celulolítica está ampliamente distribuida en el reino Fungi; se ha reportado en hongos anaerobios de los géneros Neocallimastix, Piromyces, Caecomyces, Orpinomyces y Anaeromyces, y en hongos aerobios de los géneros Bulgaria, Chaetomium, Helotium, Neurospora, Coriolus, Phanerochaete, Pleurotus, Trameres, Poria, Schizophyllum, Serpula, Aspergillus, Cladosporium, Fusarium, Geotrichum, Myrothecium, Paecilomyces, Penicillium y Trichoderma35,65. Sin embargo, estudios recientes han demostrado que los hongos pertenecientes al Phylum Ascomycota dominan, tanto en abundancia como en actividad, la comunidad microbiana responsable de la descomposición de residuos celulósicos72.
La conversión de la biomasa celulósica en azúcares fermentables a través de la hidrólisis enzimática ha sido un campo de investigación y desarrollo muy amplio. Las celulasas fúngicas se comercializan desde hace más de 30 años y han demostrado su potencial biotecnológico en varias industrias, entre las que se incluyen la de alimentos, cervecera y vinícola, la agrícola, papelera y textil, y la de detergentes55. Sin embargo, el alto coste de producción de estas enzimas y su baja eficiencia han limitado las aplicaciones industriales; es el caso de la producción de etanol a partir de celulosa, donde la hidrólisis enzimática sigue siendo una de las mayores dificultades, tanto en lo técnico como en lo económico27. Es por ello que la industria demanda enzimas más estables, más activas y más económicas. Para dar respuesta a estos requerimientos es necesario entender, entre otras cosas, los mecanismos de acción de estas enzimas, así como la regulación de su expresión en microorganismos celulolíticos. Dentro de este contexto, el presente trabajo de revisión se centra en los mecanismos de acción enzimática, los genes involucrados y los mecanismos de regulación génica para 3 de los modelos de hongos celulolíticos más estudiados: Trichoderma reesei, Aspergillus niger y Aspergillus nidulans, y para un modelo recientemente descrito, Neurospora crassa.
Estructura de la celulosaLa celulosa es un polisacárido sintetizado en gran cantidad por las plantas, constituye entre el 35 y el 50% del peso seco de estas35, y también es sintetizado por bacterias y una variedad amplia de algas37. Está constituida por moléculas de D-glucosa unidas por enlaces glucosídicos β-1,4, y se requieren 8 unidades monoméricas de glucosa para formar un producto insoluble. Dependiendo de la fuente, puede tener entre 8.000 y 15.000 unidades monoméricas por cadena37. El polisacárido está localizado en la pared celular, donde se encuentra como unidades submicroscópicas de forma alargada llamadas micelas. Estas micelas se organizan en estructuras más grandes, las microfibrillas, las cuales se empaquetan formando una estructura cristalina altamente ordenada, en la cual todos los átomos están fijos en posiciones discretas uno con respecto a otro; este empaquetamiento previene la penetración no solo de enzimas, sino de pequeñas moléculas como el agua. Esta estructura se encuentra rodeada por polisacáridos hemicelulósicos que se unen a la celulosa por puentes de hidrógeno y enlaces covalentes, haciéndola aún más resistente a la hidrólisis química y biológica35. No toda la estructura de la celulosa es cristalina, existen regiones «desordenadas», denominadas regiones amorfas, con una composición heterogénea caracterizada por una variedad de enlaces. Este arreglo asimétrico que caracteriza las regiones amorfas es crucial para la biodegradación de la celulosa38. Además de estas regiones, las fibras de celulosa contienen varios tipos de irregularidades, como torceduras y espacios, en las cuales se forman microporos y capilares lo suficientemente amplios como para permitir la penetración de moléculas relativamente grandes, incluyendo en algunos casos las enzimas celulolíticas35.
Mecanismos de acción enzimáticaLa hidrólisis enzimática de la celulosa implica la acción secuencial de un grupo de enzimas, conocidas como celulasas, que pertenecen a la superfamilia de las glicosil hidrolasas, llamadas así porque catalizan la hidrólisis del enlace glucosídico entre 2 o más hidratos de carbono o entre estos y una fracción que no sea un hidrato de carbono. Las glicosil hidrolasas han sido clasificadas en más de 100 familias, y cada una de estas (familia GH) contiene proteínas que están relacionadas por su secuencia, su estructura y, en consecuencia, por su mecanismo catalítico12,73. La arquitectura general deducida para estas enzimas presenta 2 módulos globulares independientes: un dominio catalítico, responsable de la reacción de hidrólisis per se, y un módulo de unión a la celulosa, que desempeña 3 funciones generales: la primera es mantener la enzima próxima al sustrato, la segunda es una función de reconocimiento, y, por último, una función disruptiva7,25.
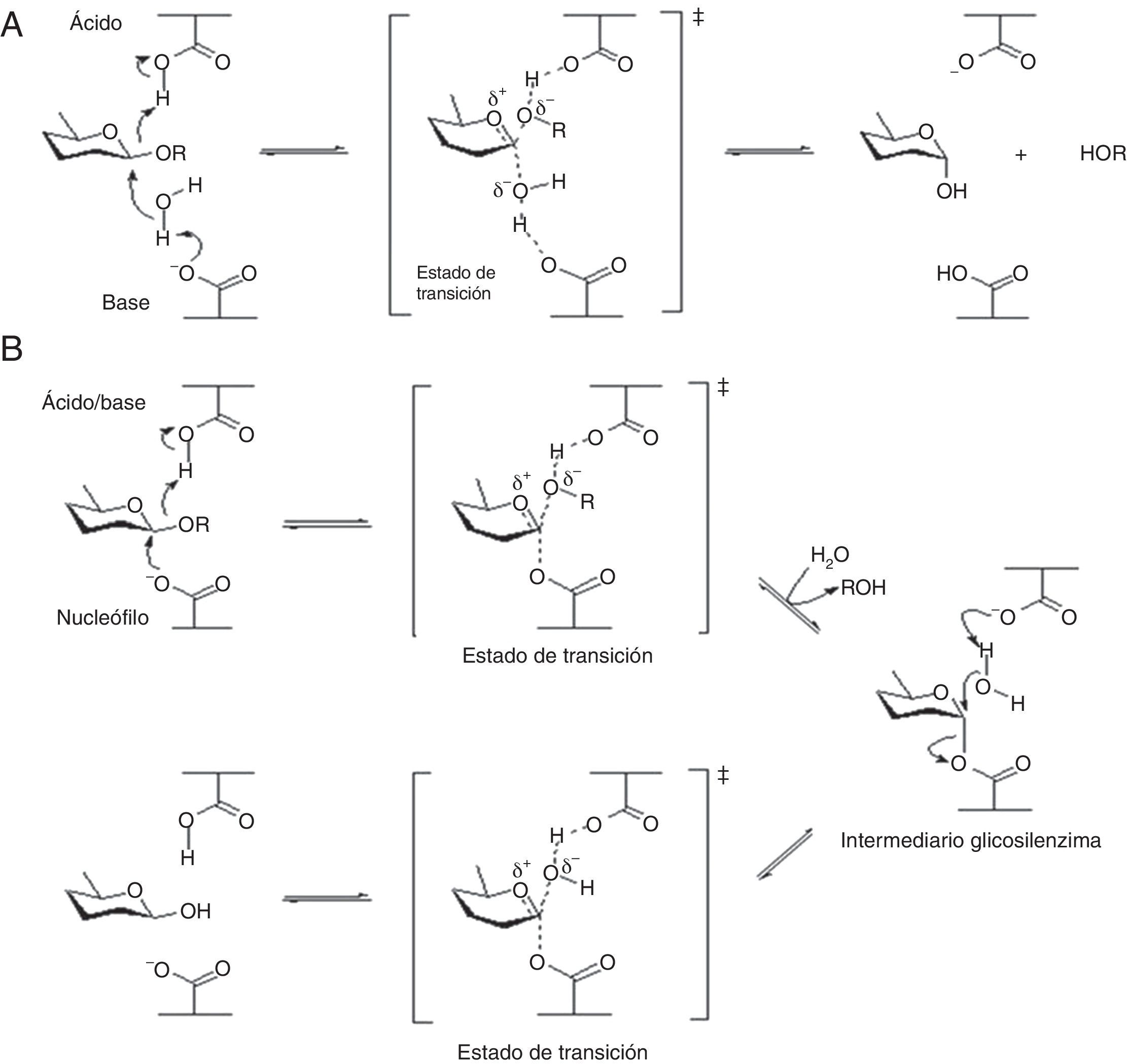
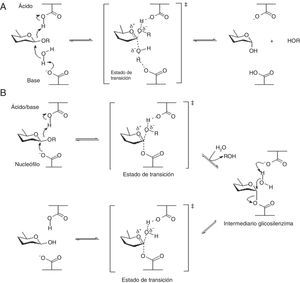
Se han descrito 2 mecanismos de hidrólisis para las glicosil hidrolasas, de conservación y de inversión de la configuración del carbono anomérico. En general, la hidrólisis del enlace glucosídico es catalizada por 2 aminoácidos en el sitio catalítico, uno ácido general (donante de protones) y otro nucleófilo/base, aspartato y glutamato, generalmente73. Dependiendo de la posición espacial de estos aminoácidos la hidrólisis se produce a través de un mecanismo o de otro. Si estos están dispuestos a una distancia de ≈5,5Å se da el mecanismo de conservación, si por el contrario esta distancia es de ≈10Å sucede el mecanismo de inversión12. En el mecanismo de conservación, la hidrólisis se da en 2 etapas. En la primera, glucosilación, el nucleófilo ataca el centro anomérico, dando como resultado la formación de una enzima glucosídica intermedia con un carácter ácido provisto por el carboxilato ácido. En la segunda, deglucosilación, el ahora desprotonado carboxilato ácido actúa como base y se une a una molécula de agua nucleofílica para hidrolizar la enzima intermedia, obteniéndose así el producto hidrolizado73 (fig. 1B). Por otro lado, en el mecanismo de inversión, la hidrólisis es alcanzada en una sola etapa, ya que debido a la mayor distancia entre los residuos catalíticos la molécula de agua puede ser acomodada entre la base y el azúcar12,73 (fig. 1A).
Mecanismos de hidrólisis enzimática de la β-glucosidasa. A. Mecanismo de inversión. B. Mecanismo de conservación. Traducida de Withers y Williams73, reproducida con permiso.
En los microorganismos que producen celulasas, estas pueden estar formando complejos unidos a la membrana celular llamados celulosomas, como en las bacterias anaerobias (especialmente, Clostridium spp.) y algunos hongos (de los géneros Neocallimastix, Piromonas y Sphaeromonas), o en un sistema enzimático no en complejo, como es el caso de los hongos aerobios38, objeto de esta revisión. De cualquier forma, el sistema de celulasas típico incluye 3 tipos de enzimas: 1) endoglucanasas o 1,4-β-D-glucano-4-glucanohidrolasas (EC.3.2.1.4); 2) exoglucanasas, que incluyen 1,4-β-D-glucano-glucanohidrolasas (celodextrinasas) (EC 3.2.1.74) y 1,4-β-D-glucano-celobiohidrolasas (celobiohidrolasas) (EC.3.2.1.91), y 3) β-glucosidasas o β-glucosidasa hidrolasas (EC.3.2.1.21)13,35,38. Las endoglucanasas cortan al azar en el interior de la celulosa amorfa, generando oligosacáridos de varias longitudes y, en consecuencia, nuevos extremos de cadena. Las exoglucanasas actúan de una manera progresiva en los extremos reductores y no reductores de las cadenas del polisacárido, liberando glucosa (glucanohidrolasas) o celobiosa (celobiohidrolasas). Las β-glucosidasas hidrolizan las celodextrinas solubles y la celobiosa a glucosa35,38.
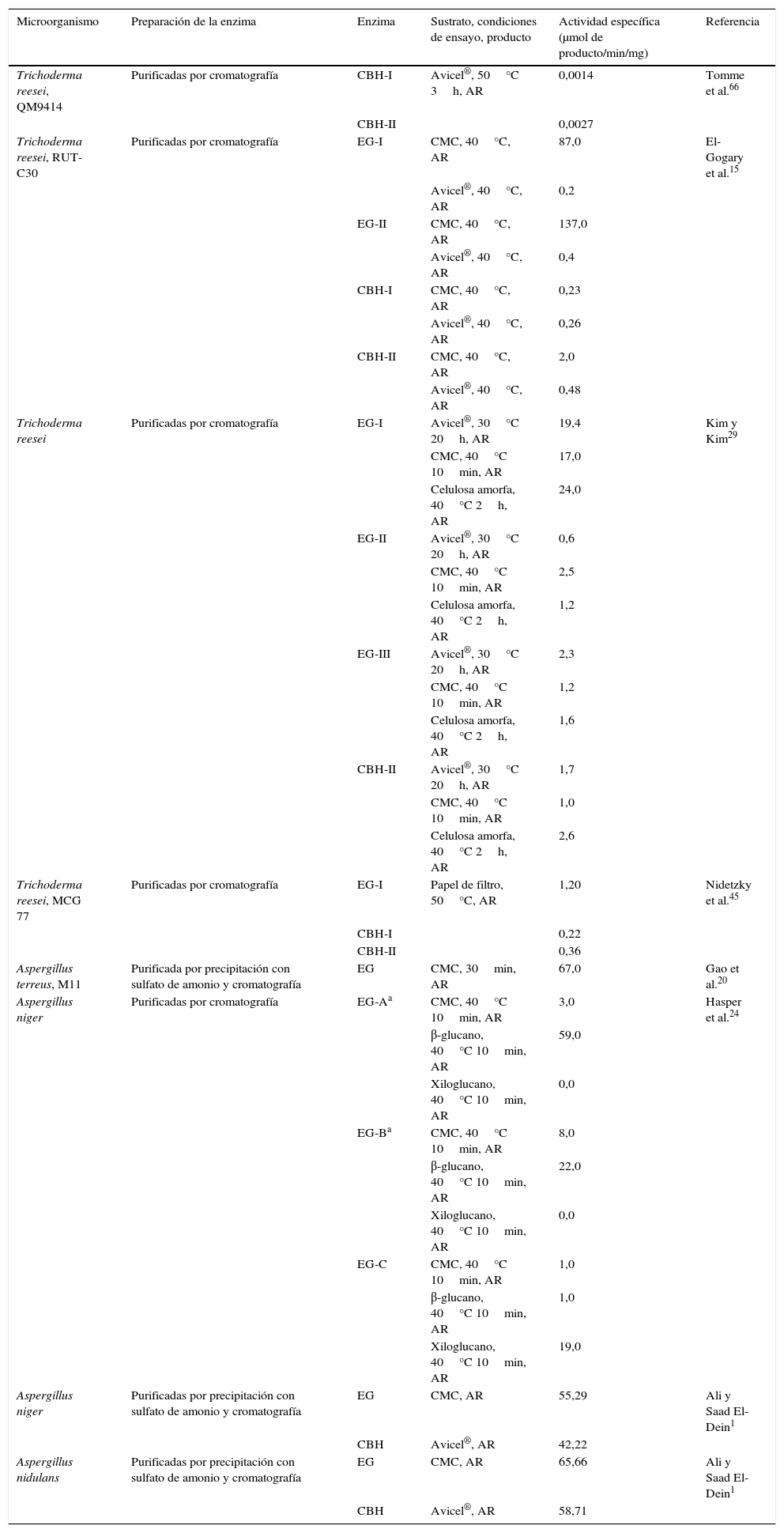
La estructura de la celulosa representa un reto para este sistema enzimático. Las características estructurales que afectan fundamentalmente la hidrólisis enzimática son la cristalinidad y el área de superficie, siendo esta última importante porque el contacto físico entre la enzima y la celulosa es un prerrequisito para la hidrólisis. En cuanto a la cristalinidad, esta afecta en la medida en que disminuye la accesibilidad de las enzimas al sustrato, de manera que la celulosa amorfa es rápidamente degradada y, a medida que aumenta el grado de cristalinidad, la celulosa comienza a ser resistente a la hidrólisis16. En la tabla 1 se presentan valores reportados de actividad específica de enzimas purificadas obtenidas a partir de T. reesei y Aspergillus spp. Al comparar los datos presentados, la primera conclusión obvia es la alta variabilidad entre la actividad de las enzimas obtenidas de microorganismos del mismo género y especie, purificadas aparentemente de la misma manera y evaluadas con los mismos sustratos. Esta variabilidad puede deberse a diferencias entre las condiciones de ensayo utilizadas por los investigadores y, probablemente, al uso de preparados con diversos grados de pureza obtenidos usando diferentes protocolos de purificación. A pesar de esta variabilidad, se puede observar una mayor actividad en sustratos con menor grado de cristalinidad (CMC, celulosa amorfa y β-glucano), y una menor actividad en sustratos con mayor grado de cristalinidad (Avicel® y papel de filtro). En estos últimos, la accesibilidad de la enzima al sustrato es el factor limitante para la hidrólisis, mientras que las propiedades de superficie pasan a un segundo plano, ya que su efecto se da únicamente en las fases iniciales de la reacción16,22.
Actividades específicas de endoglucanasas (EC.3.2.1.4) y exoglucanasas (celobiohidrolasas) (EC.3.2.1.91) reportadas para Trichoderma reesei y Aspergillus spp
| Microorganismo | Preparación de la enzima | Enzima | Sustrato, condiciones de ensayo, producto | Actividad específica (μmol de producto/min/mg) | Referencia |
|---|---|---|---|---|---|
| Trichoderma reesei, QM9414 | Purificadas por cromatografía | CBH-I | Avicel®, 50°C 3h, AR | 0,0014 | Tomme et al.66 |
| CBH-II | 0,0027 | ||||
| Trichoderma reesei, RUT-C30 | Purificadas por cromatografía | EG-I | CMC, 40°C, AR | 87,0 | El-Gogary et al.15 |
| Avicel®, 40°C, AR | 0,2 | ||||
| EG-II | CMC, 40°C, AR | 137,0 | |||
| Avicel®, 40°C, AR | 0,4 | ||||
| CBH-I | CMC, 40°C, AR | 0,23 | |||
| Avicel®, 40°C, AR | 0,26 | ||||
| CBH-II | CMC, 40°C, AR | 2,0 | |||
| Avicel®, 40°C, AR | 0,48 | ||||
| Trichoderma reesei | Purificadas por cromatografía | EG-I | Avicel®, 30°C 20h, AR | 19,4 | Kim y Kim29 |
| CMC, 40°C 10min, AR | 17,0 | ||||
| Celulosa amorfa, 40°C 2h, AR | 24,0 | ||||
| EG-II | Avicel®, 30°C 20h, AR | 0,6 | |||
| CMC, 40°C 10min, AR | 2,5 | ||||
| Celulosa amorfa, 40°C 2h, AR | 1,2 | ||||
| EG-III | Avicel®, 30°C 20h, AR | 2,3 | |||
| CMC, 40°C 10min, AR | 1,2 | ||||
| Celulosa amorfa, 40°C 2h, AR | 1,6 | ||||
| CBH-II | Avicel®, 30°C 20h, AR | 1,7 | |||
| CMC, 40°C 10min, AR | 1,0 | ||||
| Celulosa amorfa, 40°C 2h, AR | 2,6 | ||||
| Trichoderma reesei, MCG 77 | Purificadas por cromatografía | EG-I | Papel de filtro, 50°C, AR | 1,20 | Nidetzky et al.45 |
| CBH-I | 0,22 | ||||
| CBH-II | 0,36 | ||||
| Aspergillus terreus, M11 | Purificada por precipitación con sulfato de amonio y cromatografía | EG | CMC, 30min, AR | 67,0 | Gao et al.20 |
| Aspergillus niger | Purificadas por cromatografía | EG-Aa | CMC, 40°C 10min, AR | 3,0 | Hasper et al.24 |
| β-glucano, 40°C 10min, AR | 59,0 | ||||
| Xiloglucano, 40°C 10min, AR | 0,0 | ||||
| EG-Ba | CMC, 40°C 10min, AR | 8,0 | |||
| β-glucano, 40°C 10min, AR | 22,0 | ||||
| Xiloglucano, 40°C 10min, AR | 0,0 | ||||
| EG-C | CMC, 40°C 10min, AR | 1,0 | |||
| β-glucano, 40°C 10min, AR | 1,0 | ||||
| Xiloglucano, 40°C 10min, AR | 19,0 | ||||
| Aspergillus niger | Purificadas por precipitación con sulfato de amonio y cromatografía | EG | CMC, AR | 55,29 | Ali y Saad El-Dein1 |
| CBH | Avicel®, AR | 42,22 | |||
| Aspergillus nidulans | Purificadas por precipitación con sulfato de amonio y cromatografía | EG | CMC, AR | 65,66 | Ali y Saad El-Dein1 |
| CBH | Avicel®, AR | 58,71 |
AR: azúcares reductores; CBH: celobiohidrolasa o exoglucanasa; CMC: carboximetilcelulosa; EG: endoglucanasa.
Por otro lado, los sistemas de celulasas no son simplemente una aglomeración de enzimas, ya que todas ellas actúan de manera coordinada y sinérgica para una hidrólisis eficiente. Se han descrito 4 tipos de sinergia: 1) endo-exo sinergia entre endoglucanasas y exoglucanasas; 2) exo-exo sinergia entre exoglucanasas que procesan los extremos reductores y no reductores de la cadena; 3) sinergia entre exoglucanasas y β-glucosidasas que eliminan celobiosa y celodextrinas, y 4) sinergia intramolecular entre los dominios catalíticos y los dominios de unión a la celulosa35. Además, se ha reportado para T. reesei la participación en este sistema de proteínas no hidrolíticas similares a las expansinas de las plantas, las cuales debilitan las uniones no covalentes entre las fibras de celulosa sin hidrólisis, aumentando de esta manera la accesibilidad de las enzimas hidrolíticas al sustrato54. Debido a esto, los preparados comerciales de celulasas consisten en cócteles enzimáticos que contienen no solamente endocelulasas, exocelulasas y β-glucosidasas, sino también xilanasas, pectinasas, etc., dependiendo de la composición de la biomasa, a la cual serán aplicadas57. La mayoría de estos preparados son producidos a partir de T. reesei y Trichoderma longibrachiatum (tabla 2)57. Sin embargo, debido a que Trichoderma tiene baja actividad de β-glucosidasa, algunos de estos preparados consisten en mezclas de enzimas producidas por Trichoderma y otros microorganismos como A. niger y, más recientemente, especies de Penicillium23,57. La eficiencia hidrolítica de estos complejos enzimáticos depende tanto de las propiedades individuales de cada enzima (eficiencia catalítica, estabilidad a temperatura alta y pH ácido), como de su proporción en la mezcla, lo que influye en el tipo de sinergia que se establece23,69.
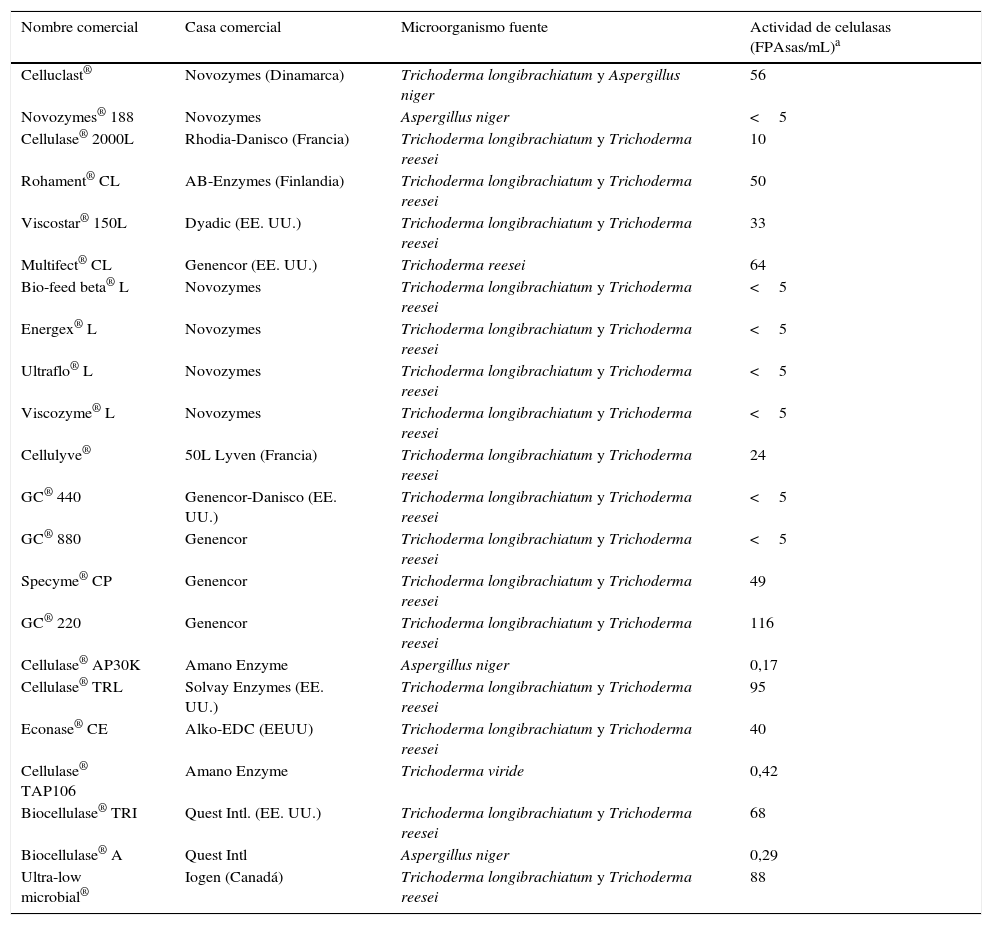
Celulasas comerciales, compañías que las producen, microorganismos fuente y actividad enzimática reportada
| Nombre comercial | Casa comercial | Microorganismo fuente | Actividad de celulasas (FPAsas/mL)a |
|---|---|---|---|
| Celluclast® | Novozymes (Dinamarca) | Trichoderma longibrachiatum y Aspergillus niger | 56 |
| Novozymes® 188 | Novozymes | Aspergillus niger | <5 |
| Cellulase® 2000L | Rhodia-Danisco (Francia) | Trichoderma longibrachiatum y Trichoderma reesei | 10 |
| Rohament® CL | AB-Enzymes (Finlandia) | Trichoderma longibrachiatum y Trichoderma reesei | 50 |
| Viscostar® 150L | Dyadic (EE. UU.) | Trichoderma longibrachiatum y Trichoderma reesei | 33 |
| Multifect® CL | Genencor (EE. UU.) | Trichoderma reesei | 64 |
| Bio-feed beta® L | Novozymes | Trichoderma longibrachiatum y Trichoderma reesei | <5 |
| Energex® L | Novozymes | Trichoderma longibrachiatum y Trichoderma reesei | <5 |
| Ultraflo® L | Novozymes | Trichoderma longibrachiatum y Trichoderma reesei | <5 |
| Viscozyme® L | Novozymes | Trichoderma longibrachiatum y Trichoderma reesei | <5 |
| Cellulyve® | 50L Lyven (Francia) | Trichoderma longibrachiatum y Trichoderma reesei | 24 |
| GC® 440 | Genencor-Danisco (EE. UU.) | Trichoderma longibrachiatum y Trichoderma reesei | <5 |
| GC® 880 | Genencor | Trichoderma longibrachiatum y Trichoderma reesei | <5 |
| Specyme® CP | Genencor | Trichoderma longibrachiatum y Trichoderma reesei | 49 |
| GC® 220 | Genencor | Trichoderma longibrachiatum y Trichoderma reesei | 116 |
| Cellulase® AP30K | Amano Enzyme | Aspergillus niger | 0,17 |
| Cellulase® TRL | Solvay Enzymes (EE. UU.) | Trichoderma longibrachiatum y Trichoderma reesei | 95 |
| Econase® CE | Alko-EDC (EEUU) | Trichoderma longibrachiatum y Trichoderma reesei | 40 |
| Cellulase® TAP106 | Amano Enzyme | Trichoderma viride | 0,42 |
| Biocellulase® TRI | Quest Intl. (EE. UU.) | Trichoderma longibrachiatum y Trichoderma reesei | 68 |
| Biocellulase® A | Quest Intl | Aspergillus niger | 0,29 |
| Ultra-low microbial® | Iogen (Canadá) | Trichoderma longibrachiatum y Trichoderma reesei | 88 |
Modificada de Singhania et al. (2010)57.
T. reesei (anamorfo de Hypocrea jecorina) ha sido el modelo de microorganismo celulolítico por excelencia. Fue aislado en las islas Salomón durante la Segunda Guerra Mundial como el agente causal de la infección masiva del material a base de algodón del ejército de los Estados Unidos. Inicialmente fue identificado como Trichoderma viride, codificado como QM6a y conservado en el Centro de Investigación y Desarrollo del Ejército de los Estados Unidos en Natick, Massachusetts32,48. Su potencial como productor de celulasas fue reconocido por Mandels y Reese en 195741, más tarde fue clasificado como una nueva especie y renombrado como T. reesei en honor al investigador del laboratorio de Natick, Elwyn T. Reese48. Desde entonces, este microorganismo ha sido objeto de numerosas investigaciones. En Japón, por ejemplo, la obtención de mutantes hiperproductores de celulasas es parte de un proyecto nacional para la generación de combustibles alternativos al petróleo desde 197046. Como resultado de este proyecto se han obtenido 2 mutantes importantes producto de una mutagénesis clásica, KDG-12 y PC3-7, que muestran una alta productividad de celulasas, resistencia a la represión catabólica por glucosa y alta inducibilidad por sorbosa28. Otro mutante hiperproductor de celulasas muy utilizado es RUT-C30, obtenido en la Universidad Rutgers, en Nueva Jersey, en 1977. Esta cepa fue generada mediante la combinación de mutagénesis clásica y radiación UV, y es capaz de producir de 15 a 20 veces más celulasas que QM6a, además de evitar la represión catabólica por glucosa48. Estos mutantes no solo han servido para la producción de enzimas celulolíticas a escala industrial, sino que también han sido utilizados como modelos para entender la regulación de la expresión de estas enzimas en T. reesei.
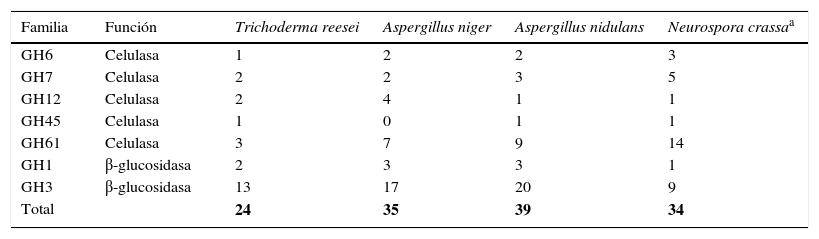
El genoma de T. reesei tiene un contenido de guanina-citosina del 52%, un tamaño de 33,9Mb y 9.129 genes que corresponden al 40,4% codificante. La longitud promedio de un gen es de 1.793pb, con 3,1 exones por gen (longitud promedio del exón, 508pb; longitud promedio del intrón, 120pb). Dentro de este genoma se han identificado 200 genes que codifican para glicosil hidrolasas42, de los cuales 2 codifican para celobiohidrolasas (EC.3.2.1.91), 7 para endoglucanasas (EC.3.2.1.4) y 15 para β-glucosidasas (EC.3.2.121) (tabla 3), cuya expresión es dirigida simultáneamente por inductores específicos, como la celulosa o subproductos de esta, y reprimidos simultáneamente por altos niveles de glucosa17,42,46.
Número de genes que codifican para enzimas celulolíticas en Trichoderma reesei y en otros modelos
| Familia | Función | Trichoderma reesei | Aspergillus niger | Aspergillus nidulans | Neurospora crassaa |
|---|---|---|---|---|---|
| GH6 | Celulasa | 1 | 2 | 2 | 3 |
| GH7 | Celulasa | 2 | 2 | 3 | 5 |
| GH12 | Celulasa | 2 | 4 | 1 | 1 |
| GH45 | Celulasa | 1 | 0 | 1 | 1 |
| GH61 | Celulasa | 3 | 7 | 9 | 14 |
| GH1 | β-glucosidasa | 2 | 3 | 3 | 1 |
| GH3 | β-glucosidasa | 13 | 17 | 20 | 9 |
| Total | 24 | 35 | 39 | 34 | |
Fuente: Kubicek31.
Encontrar la respuesta a la pregunta: «¿Cómo la celulosa, siendo un sustrato insoluble, es capaz de inducir la expresión de enzimas celulolíticas?» ha sido objeto de numerosas investigaciones. La primera hipótesis sobre la que se trabajó es que la baja expresión basal de celulasas hace que se libere glucosa y celobiosa y que estos subproductos solubles actúan como inductores de la actividad enzimática. Al respecto, Mandels y Reese encontraron en 1960 que la glucosa no es un inductor, muy al contrario, actúa como un inhibidor, y que la celobiosa, aunque induce, lo hace muy pobremente40. Estos mismos investigadores, en 1962 reportaron que la soforosa (β-D-glucosa-[1,2]-D-glucosa), una impureza de la glucosa obtenida de la hidrólisis ácida del almidón, induce 2.500 veces más la expresión de celulasas comparada con la celobiosa, que parecía ser el inductor natural lógico39. Esta observación fue confirmada por Sternberg y Mandels, quienes estudiaron, además, los factores que afectan la inducción, encontrando que la cinética, así como el efecto del pH y la temperatura sobre este, son muy similares a lo encontrado cuando la inducción se realiza con celulosa, evidenciando que la soforosa podría ser el inductor natural58. Esto último se confirmó en estudios posteriores, en los que se demostró la formación de soforosa in vitro en suspensión de células lisadas de T. reesei68 y la presencia de una β-glucosidasa constitutiva localizada en la membrana plasmática, que podría ser la responsable de dicha formación67.
Aunque se había identificado la soforosa como el inductor natural de la síntesis de celulasas, seguía sin respuesta la pregunta de cómo se induce esta síntesis. En 1987, Kubicek estudió la inducción de endoglucanasas en conidios, micelio y protoplastos de T. reesei y encontró que la celulosa induce la síntesis de endoglucanasas solamente en conidios, mientras que otros celooligosacáridos son capaces de inducir esta síntesis en micelio, conidios y protoplastos. Además, Kubicek encontró que cuando se inhibe la β-glucosidasa localizada en la membrana plasmática, se bloquea la síntesis de endoglucanasas cuando se utiliza celulosa o celooligosacáridos como inductores, para conidios o protoplastos y micelio, respectivamente, mientras que la inducción por soforosa no se ve afectada30. Este mismo investigador, en 1988 reportó la presencia de celobiohidralasas unidas a la superficie de los conidios, concluyendo así que estos expresan todas las enzimas del complejo celulolítico33. Recientemente, estudios de expresión génica mediante microarrays permitieron comparar la expresión de 900 genes durante la transición de hifa a conidio, encontrándose aumentada en este último la transcripción de los genes involucrados en transporte, síntesis de metabolitos secundarios, lipasas, proteasas y, particularmente, celulasas y hemicelulasas, confirmando de esta manera el papel de los conidios en la hidrólisis de celulosa y hemicelulosa43.
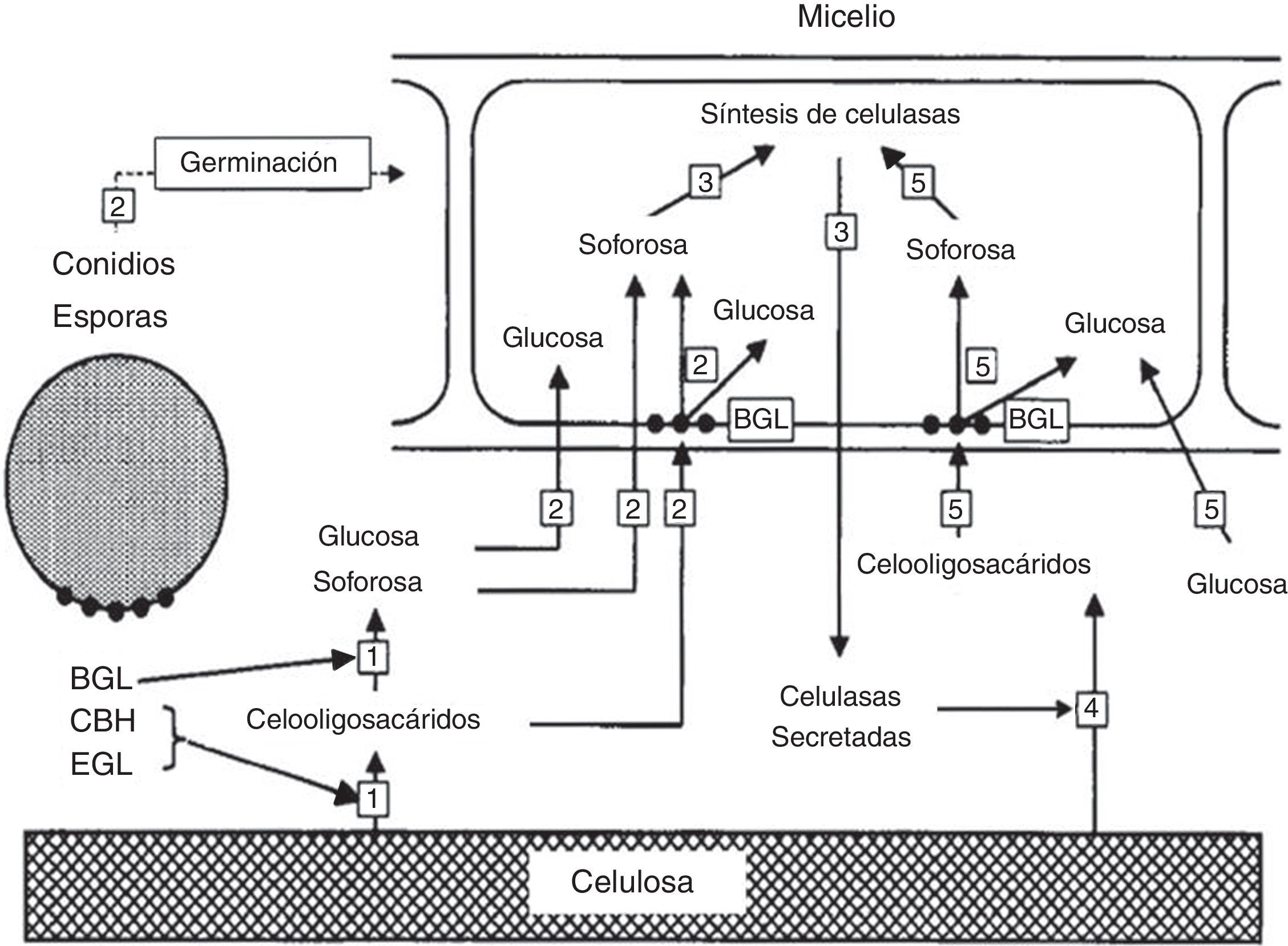
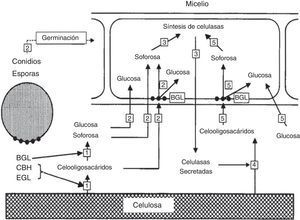
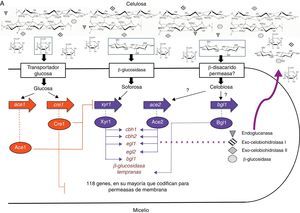
Lo anteriormente expuesto se puede resumir en el modelo propuesto por Suto y Tomita en 2001, que comprende 6 etapas: primera, los conidios entran en contacto con la celulosa y las celulasas en su superficie la degradan a celooligosacáridos, que son luego hidrolizados a glucosa y transglucosilados a soforosa por una β-glucosidasa constitutiva; segunda, los conidios germinan y la glucosa como fuente de carbono y la soforosa como inductor entran en la célula. La β-glucosidasa, unida a la membrana plasmática, convierte los celooligosacáridos en soforosa y glucosa; tercera, la soforosa induce la síntesis de celulasas que son secretadas fuera de la célula; cuarta, las celulasas secretadas degradan la celulosa causando un aumento en la cantidad de celooligosacáridos y de glucosa; quinta, la glucosa es asimilada, los celooligosacáridos son hidrolizados a glucosa y transglucosilados para formar soforosa; y sexta, el ciclo continúa hasta que el sustrato se agota64 o el sistema es reprimido por represión catabólica, como se explicará más adelante (fig. 2).
Modelo de inducción de celulasas en Trichoderma reesei. Los números se corresponden con los usados en la explicación en el texto.
BGL: β-glucosidasa; CBH: 1,4-β-D-glucano celobiohidrolasa; EGL: endo-1,4-β-D-glucanasa.
Traducida de Suto y Tomita64, reproducida con permiso.
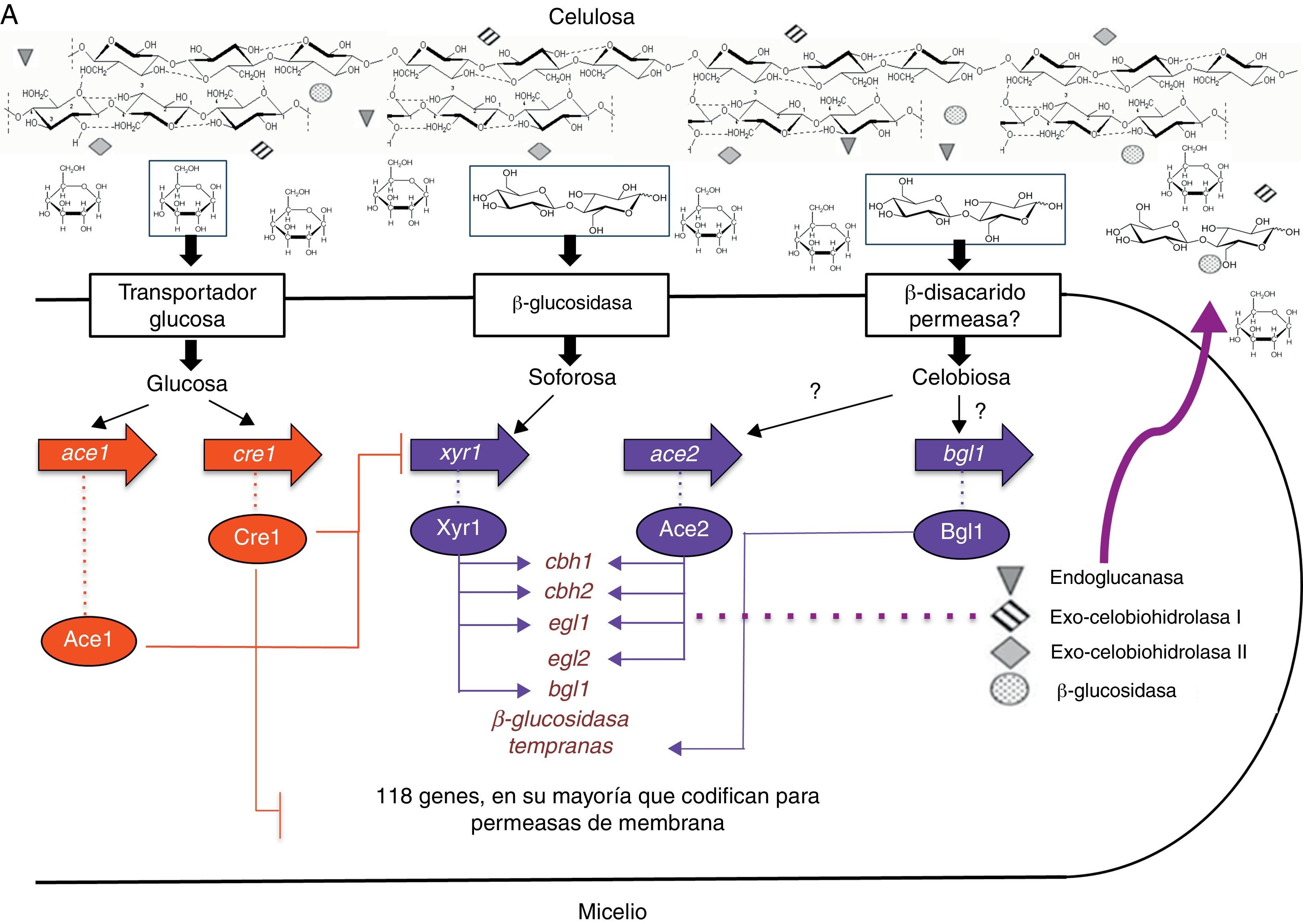
Aunque ya se ha contestado parte de la pregunta inicial, aún no se ha respondido cómo es regulado este sistema. A este respecto, la presencia obligatoria de un inductor (en este caso, soforosa) para la expresión de estas enzimas implica una regulación a nivel de promotor. Hasta la fecha se han identificado 4 activadores transcripcionales (Xyr1, Ace2, Bgl1 y el complejo HAP2/3/5) y 2 represores (Ace1 y el represor catabólico por carbono Cre1)32,46 (tabla 4).
Genes reportados como reguladores (positivos o negativos) de enzimas celulolíticas en Trichoderma reesei y en otros modelos
| Gen | Función en Trichoderma reesei | Genes ortólogos reportados en otros modelos | |
|---|---|---|---|
| Aspergillus niger/nidulans | Neurospora crassa | ||
| xyr1 | Activador de transcripción de xyn1, xyn2, bxl1, cbh1, cbh2, egl1, and bgl136,50,61 | xlnR70,71 | |
| ace2 | Activador de transcripción de cbh1, cbh2, egl1 y egl24 | ||
| bgl1 | Activador de transcripción temprana de genes de β-glucosidasas46 | ||
| ace1 | Regulador negativo3,53 | ||
| cre1 | Regulador negativo10,26,44,60 | creA14,52 | cre163 |
| cre2 | creB34 | clr28 | |
| cre3 | creC34 | ||
| creD6 | |||
| clr18 | |||
Xyr1 es una proteína de unión al ADN con un cluster binuclear de cinc que se encuentra exclusivamente en hongos. Esta proteína se une al motivo GGCTA ubicado como una repetición invertida separada por 10pb (5’-GGCTAAATGCGACATCTTAGCC-3’) en la región promotora50. Inicialmente fue identificado como inductor de xilanasas; sin embargo, Stricker et al., en 2006, demostraron que la deleción de xyr1 en T. reesei no solamente restringe fuertemente la utilización del xilano, sino que también elimina la inducción de celulasas por celulosa y soforosa, indicando que Xyr1 es un activador general y esencial de la transcripción, que gobierna la expresión del sistema enzimático celulolítico (cbh1, cbh2, egl1 y bgl1) y participa en la regulación del sistema xilanolítico (xyn1 y xyn2) en T. reesei36,61. Aro et al., en 2001, aislaron un segundo activador de la expresión de celulasas, Ace2. Al igual que Xyr1, es una proteína de unión a ADN con un cluster binuclear de cinc, se une a la secuencia 5’-GGCTAATAA-3’ del promotor del gen cbh1 y regula los genes cbh1, cbh2, egl1 y egl2. La deleción de ace2 lleva a una disminución en la cantidad de ARNm de las celobiohidrolasas i y ii y de las endoglucanasas i y ii, y a una disminución en la actividad de celulasas totales, entre el 30 y 70%. Al contrario de lo que podría esperarse, la inducción de celulasas por soforosa no se ve afectada por la deleción de ace2, lo que indica que para la inducción por celulosa y soforosa se emplean al menos parcialmente diferentes mecanismos moleculares4. Recientemente se describió otro activador transcripcional, denominado Bgl1, con la misma estructura mencionada para los 2 anteriores. Bgl1 regula la transcripción temprana de genes específicos que codifican para β-glucosidasas46. Por otro lado, Zeilinger et al. (1988 y 2001) encontraron que la expresión del promotor cbh2 depende de la unión del complejo proteico HAP2/3/5 a la caja CCAAT, un elemento cis encontrado en las regiones promotoras de un gran número de genes eucariotas. Al parecer, el complejo proteico se encarga de la desestabilización de la cromatina necesaria para la activación de la transcripción74,75.
Los 2 reguladores negativos descritos hasta ahora son Cre1 y Ace1. Cre1 fue descrito por primera vez en 1995 por Strauss y et al. como una proteína de unión a ADN con la estructura típica de «dedo de cinc", Cys2His2, y algunas regiones ricas en prolina y aminoácidos básicos que pueden estar involucradas en la interacción con otras proteínas. Cre1 se une a la secuencia 5’-GCGGAG-3’, probablemente como un complejo proteico60, y se ha determinado que la fosforilación de la serina de la posición 241 es fundamental para la unión de Cre1 a la secuencia blanco10. La dinámica de represión catabólica de Cre1 ha sido mejor comprendida gracias al estudio del mutante RUT-C30, en el cual cre1 aparece truncado y solo codifica para una de las 2 regiones de «dedos de cinc» de la proteína Cre1. De esta manera, T. reesei RUT-C30 produce celulasas y hemicelulasas en un medio que contiene glucosa, y cuando se complementa la mutación con el gen cre1 de la cepa silvestre se restaura el fenotipo de represión por glucosa26. Nakari-Setälä et al. (2009) encontraron que el papel de Cre1 va más allá de la «simple» desrepresión por glucosa al demostrar que la deleción de cre1 en una cepa silvestre lleva al incremento tanto en la actividad como en la cantidad de ARNm de celulasas en un medio con glucosa, resultado esperado por la liberación de la represión catabólica. Sin embargo, también observaron que la producción de celulasas en el medio con glucosa fue mucho más baja que en un medio con celulosa para la cepa deficiente en cre1, así como un gran aumento en la producción de proteínas en el medio con celulosa, evidenciando que cre1 juega un papel en la modulación de la expresión de genes de celulasas bajo condiciones de inducción44.
Saloheimo et al., en 2000, aislaron un gen que codifica para otro factor de transcripción de genes de celulasas, al que denominaron Ace1. Esta proteína contiene 3 motivos «dedo de cinc» del tipo Cys2His2, y establecieron que in vitro se une al promotor cbh153. Posteriores estudios mostraron que la deleción de este gen aumenta de 2 a 30 veces la transcripción de los genes cbh1, cbh2, egl1 y egl2 y, en consecuencia, aumenta la habilidad del hongo para degradar la celulosa, indicando que Ace1 es un regulador negativo y que este mecanismo es independiente de Cre13.
Con el advenimiento de las «ómicas», se han realizado nuevos estudios sobre estos «viejos modelos» intentando dar una visión más holística. En 2011, Portnoy et al. demostraron que el papel regulatorio de Cre1 es mucho más complejo de lo que se había reportado hasta la fecha. Estos investigadores identificaron 207 genes regulados por Cre1, de los cuales 118 son regulados negativamente por este. Dentro de este grupo, un alto porcentaje corresponde a genes que codifican para permeasas de membrana, lo que indica que la represión catabólica actúa preferencialmente en la entrada de sustratos a la célula y constituye un mecanismo por el que los hongos pueden recuperar incluso trazas de fuentes de carbono a una velocidad alta. También encontraron que las permeasas que transportan compuestos de nitrógeno son reguladas por Cre1, hecho que evidencia que las proteínas y sus productos de degradación son sustratos preferidos para un metabolismo rápido de los hidratos de carbono49. Estudios del secretoma de T. reesei cultivado en celulosa como fuente de carbono han permitido identificar, además de celulasas y hemicelulasas, lacasas, glioxal oxidasas (una metaloenzima importante en la degradación de lignina), peptidasas y amidasas, lo que lleva a especular que el rompimiento de la celulosa por T. reesei puede ser asistido por oxidasas31, hecho que debe ser probado y que cambiaría el paradigma de los mecanismos de hidrólisis de la celulosa. Otro dato interesante es la detección de enzimas intracelulares (tales como deshidrogenasas) en los filtrados de los cultivos, indicando que la secreción de estas enzimas está acompañada de autolisis o fragmentación del micelio, si bien estos hallazgos aún carecen de mayor evidencia experimental31.
Regulación en otros modelosAspergillus niger, Aspergillus nidulansA. niger es un microorganismo muy utilizado en biotecnología dada su habilidad de secretar grandes cantidades de una gran variedad de ácidos orgánicos y de enzimas necesarias para la hidrólisis de biopolímeros, entre ellas, celulasas. El genoma de A. niger está organizado en 8 cromosomas, con un tamaño total de 33,9Mb, un contenido de guanina-citosina del 50,4% y 14.165 genes que codifican para proteínas, de los cuales 35 codifican para celulasas (tabla 3). Cada gen tiene una longitud promedio de 1.572pb y 2,57 intrones (tamaño promedio de un intrón: 97pb; tamaño promedio de un exón: 370pb)47. A. nidulans es considerado un microorganismo modelo y ha sido utilizado en el estudio de la fisiología celular en eucariotas en temas tales como regulación del metabolismo, desarrollo, control del ciclo celular, estructura de la cromatina y función del citoesqueleto, entre otros. Al igual que en A. niger, el genoma de A. nidulans está organizado en 8 cromosomas, tiene un tamaño total de 30,1Mb, un contenido de guanina-citosina del 50% y 9.541 genes que codifican para proteínas, de los cuales 39 codifican para celulasas (tabla 3), cada gen con una longitud promedio de 1.547pb y 3,6 exones (longitud promedio de un intrón: 97pb; longitud promedio de un exón: 370pb)19.
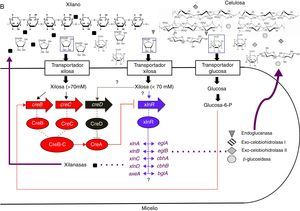
El primer reporte de un activador transcripcional de enzimas extracelulares en hongos filamentosos fue hecho para A. niger. En 1998, Van Peij et al.. identificaron una proteína de 875 aminoácidos con un cluster binuclear de cinc que se une a la secuencia 5’-GGCTAAA-3’ que aparece por triplicado en el promotor xlnA, a la que denominaron XlnR. Esta proteína activa la transcripción de los genes del sistema xilanolítico xlnA, xlnB, xlnC, xlnD y axeA, así como también la transcripción de genes que codifican para endoglucanasas, eglA y eglB, y celobiohidrolasas, eg1A y eg1B; indicando una posible coordinación en la regulación de las xilanasas y celulasas21,70,71. Aunque el gen xlnR es ortólogo de xyr1 de T. reesei, el mecanismo de regulación es diferente, mientras que en T. reesei Xyr1 induce la expresión de celulasas y algunas xilanasas a través de la inducción por soforosa39,40,58, en A. niger la transcripción de eglA, eglB, cbhA y cbhB es dirigida por XlnR por inducción de D-xilosa y no por soforosa u otro producto de degradación de la celulosa21. Al igual que en T. reesei, en este modelo se asume que una limitación por carbono lleva a una condición de desrepresión por el catabolito, y en este estado los azúcares monoméricos, D-xilosa para A. niger, o sus derivados, y soforosa para T. reesei, pueden actuar como inductores; la naturaleza de este azúcar monomérico determina el sistema enzimático que es inducido62. En A. niger el activador es expresado en presencia de xilano y lleva a una coutilización de la celulosa; en ausencia de xilano este sistema podría no ser activado o serlo pobremente, lo que conlleva un crecimiento reducido cuando la celulosa es la única fuente de carbono9.
En A. niger, la expresión de genes xilanolíticos y celulolíticos es fuertemente reprimida por glucosa, fructosa y D-xilosa. Esta represión es mediada fundamentalmente por creA, que es el ortólogo de cre1 en T. reesei14,52. Ahora bien, si la D-xilosa es el inductor, ¿cómo puede ser al mismo tiempo el represor? De Vries et al. (1999) evaluaron esta dinámica comparando la expresión de genes xilanolíticos en una cepa silvestre y una mutante para creA y encontraron que el nivel de expresión de estos genes depende del balance entre la inducción por XlnR y la represión por CreA. La D-xilosa es liberada gradualmente del xilano, lo que lleva a una baja concentración de D-xilosa en el medio. Bajo estas condiciones la represión por D-xilosa mediada por CreA es baja y se detectan niveles altos de expresión. Cuando los niveles de D-xilosa en el medio alcanzan concentraciones de 70mM, la represión a través de CreA juega un papel más importante, resultando en una disminución de los niveles de expresión14. El mecanismo de represión mediado por CreA ha sido estudiado más detalladamente en A. nidulans, en el cual se han encontrado al menos 3 mecanismos involucrados: 1) la activación de la expresión del gen creA dependiente de monosacáridos; 2) autorrepresión del gen creA, dependiente de la formación de glucosa-6-P, y 3) regulación de la función de CreA por modificaciones traduccionales o postraduccionales11,59, las cuales pueden ser dadas por un mecanismo de ubiquitinación-desubiquitinación. Respecto a este mecanismo, los genes creB, creC34y creD6 se han aislado en A. nidulans. El primero codifica para una enzima desubiquitinante que forma un complejo in vivo con la proteína codificada por creC. Se ha propuesto que este complejo elimina la ubiquitina de CreA, modificando o estabilizando la proteína34, de manera que cumpla su función; de este hecho se deduce que mutaciones en los genes creB y creC llevan a una parcial desregulación de la represión por carbono32. Por otra parte, CreD contiene dominios arrestina y motivos PY altamente similares a las proteínas de Saccharomyces cerevisiae Rod1p y Ros3p, las cuales interactúan con la ubiquitina ligasa Rsp5p, indicando el posible rol de CreD en la ubiquitinación de CreA6.
Neurospora crassaN. crassa fue aislado por primera vez en 1843 como un contaminante en una cervecería en París, y desde 1940 ha sido un microorganismo modelo para estudios genéticos, bioquímicos y de biología de hongos51. Aunque su capacidad de degradar celulosa se conoce desde hace unos 30 años65, relativamente pocos estudios se han publicado respecto a la regulación de este sistema, por lo que se puede considerar un «nuevo modelo» en este tema específico. N. crassa tiene 7 cromosomas y las características más importantes de su genoma son: contenido de guanina-citosina del 50%, un tamaño de 38,7Mb y 10.082 genes que corresponden al 38,5% codificante. La longitud promedio de un gen es de 1.673pb, con un promedio de 1,7 intrones por gen (longitud promedio de un intrón: 134 nucleótidos)18. Se ha predicho que el genoma de N. crassa contiene 171 genes que codifican para glucosilhidrolasas, un 15,5% menos que T. reesei; sin embargo, de estos genes, 34 codifican para celulasas (tabla 3), un 42% más que T. reesei.
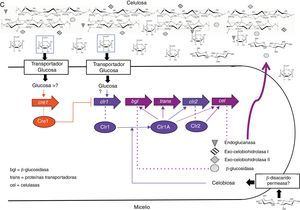
A diferencia de los 2 modelos presentados hasta ahora (T. reesi y A. niger/A. nidulans), poco se conoce acerca de la inducción y regulación de celulasas en N. crassa. El primer reporte fue hecho por Tian et al. en 2009, quienes mediante el uso de microarrays identificaron 114 genes, predominantemente involucrados en el metabolismo del carbono, que mostraron diferencias en la expresión cuando N. crassa fue cultivada sobre un sustrato lignocelulósico (Miscanthus) y sobre celulosa cristalina (Avicel®), en comparación con el crecimiento sobre sacarosa. Adicionalmente, fueron identificadas 22 proteínas, entre estas 9 de las 24 celulasas predichas, 5 hemicelulasas, 5 proteínas con actividad predicha sobre hidratos de carbono y 2 proteínas con función desconocida65. El hecho de que se expresen hemicelulasas sobre celulosa induce a especular que la celulosa podría ser el inductor primario, así como en T. reesei, en el que la inducción por celulosa induce también la expresión de hemicelulasas y xilanasas36,61. Sin embargo, esta hipótesis necesita ser comprobada. Por otra parte, en dicho estudio se identificó la celobiohidrolasa i como la proteína extracelular producida mayormente durante el crecimiento en Avicel® y en Miscanthus, y que la deleción del gen cbh1 causa deficiencias severas en el crecimiento sobre estos sustratos65, a diferencia de T. reesei, en el que el fenotipo más severo es causado por la deleción de cbh256.
Sun y Glass, en 2011, evaluaron el nivel de expresión de genes y la actividad de celulasas en un mutante para el ortólogo de cre1/creA en N. crassa. Cuando este mutante se sembró en un medio con celulosa cristalina (Avicel®) como única fuente de carbono, mostró una mayor velocidad en el consumo del sustrato en comparación con la cepa silvestre (3-4 frente a 5-6 días), el 30% más de proteínas secretadas al medio y el aumento del 50% en la actividad de endoglucasas, lo que indica el rol de cre1 en la represión de la expresión de celulasas63, al igual que sus ortólogos en T. reesei10,26,44,60 y en A. niger14,52. En este mismo estudio se identificaron 9 genes, blancos directos de Cre1, entre los que se encuentran el ortólogo de amyA de Aspergillus oryzae, involucrado en la degradación del almidón, el gen cbh1, 2 genes que codifican para xilanasas, un gen que codifica para una glucosidasa y un gen para un transportador de monosacáridos, lo que evidencia que Cre1 puede regular directamente algunos genes involucrados en el transporte de azúcares, además de genes que codifican para enzimas asociadas a la utilización de diversas fuentes de carbono63.
Recientemente se reportaron 2 nuevos factores de transcripción en N. crassa, requeridos para el crecimiento y la actividad enzimática sobre celulosa, pero no requeridos sobre hemicelulosa o xilano. Estos son clr1 y crl2, que codifican para factores de transcripción de la superfamilia del cluster binuclear de cinc específicos de hongos. Estos fueron identificados mediante la selección de mutantes con crecimiento deficiente en celulosa cristalina (Avicel®). Estos investigadores evaluaron 269 mutantes de N. crassa, cada uno de ellos con deleción de un gen para un posible factor de transcripción, incluyendo los ortólogos de xlnR/xyr1, ace1 y hap2, los cuales, en contra de lo que se esperaba, no mostraron ninguna diferencia en el crecimiento sobre Avicel® en comparación con la cepa silvestre8. De estos resultados se puede inferir que el homólogo de XlnR/Xyr1 no es necesario para la expresión de genes de celulasas en N. crassa, como sí lo es para A. niger21,70,71 y T. reesei36,61, lo que evidencia que las diferentes especies de hongos han desarrollado mecanismos diferentes en respuesta a distintos inductores, provenientes de fuentes complejas como la celulosa o la hemicelulosa. Por otra parte, Clr1 promueve la expresión de genes de celulasas, específicamente β-glucosidadas, y otros necesarios para la utilización de la celobiosa, así como la expresión de clr2, mientras que Clr2 induce la expresión de genes de otras celulasas y hemicelulasas, posiblemente formando un heterocomplejo con la molécula activa de Clr18.
Similar a lo publicado por Portnoy et al. (2011) para T. reesei49, la deleción de clr1 y clr 2 en N. crassa tiene efecto sobre un gran número de genes, indicando lo complejo de estos sistemas. Trescientos treinta y tres genes presentan expresión diferencial en estos mutantes, de los cuales 212 presentaron expresión aumentada en Avicel®, entre los que se encuentran 17 de 24 celulasas predichas, 11 de 19 hemicelulasas predichas, 22 de enzimas con actividad sobre polisacáridos, 56 de proteínas de la vía secretoria, 13 transportadores, 3 componentes del complejo de traslocación del retículo endoplasmático, 2 retículo endoplasmático disulfuro isomerasas, 2 proteínas chaperonas (HSP70, calreticulina), 7 enzimas citosólicas con actividad sobre disacáridos y un homólogo a xyr18. Se observa para N. crassa, al igual que para T. reesei, un gran número de genes que codifican para proteínas con funciones de secreción; ello evidencia que la modificación de estas vías es requerida para el incremento de la secreción de proteínas que acompañan el metabolismo de la celulosa.
Conclusiones y perspectivasA pesar de la abundancia de la celulosa, solamente un pequeño porcentaje de microorganismos son capaces de degradarla completamente; uno de los más importantes grupos corresponde a los hongos filamentosos. Dentro de este grupo, T. reesei, A. niger, A. nidulans y, recientemente, N. crassa han sido extensamente estudiados con el fin de comprender no solamente los mecanismos de hidrólisis enzimática, sino también la regulación de la expresión de los genes que codifican para estas enzimas. Los resultados de estas investigaciones muestran que los mecanismos de hidrólisis enzimática son muy similiares en estos microorganismos, no así los mecanismos de regulación, en los que se encuentran similitudes y diferencias. Una de las mayores similitudes entre T. reesei y A. niger/A. nidulans es la presencia de un factor transcripcional general (Xyr1/XlnR) responsable de la activación de la transcripción del sistema bajo condiciones de inducción. Sin embargo, esta condición de inducción es diferente: mientras que en Aspergillus la mayoría de celulasas son correguladas con las xilanasas a partir de una sola molécula inductora (D-xilosa), en T. reesei la inducción se da por soforosa o celobiosa de una manera independiente de las xilanasas. Además, en T. reesei se han identificado otros reguladores positivos, Ace2 y Bgl1, cuyos ortólogos no han sido reportados en Aspergillus (fig. 3A y B). Por otra parte, el ortólogo de xyr1/xlnR en N. crassa no es necesario para la expresión de celulasas en este microorganismo (fig. 3C), lo que demuestra que las especies de hongos han desarrollado diferentes mecanismos en respuesta a los mismos inductores.
Otra característica en común en estos sistemas es el fenómeno de represión catabólica, en donde el producto final de la hidrólisis enzimática es el directo responsable de la regulación negativa a nivel transcripcional. Este mecanismo evita que el hongo sintetice una cantidad excesiva de celulasas cuando existe disponibilidad de otras fuentes más fácilmente asimilables. En los 3 modelos estudiados, T. reesei, A. niger/A. nidulans y N. crassa, los genes ortólogos cre1/creA son los responsables de dicha regulación (fig. 3A, B y C). Aún no está del todo claro el mecanismo por el cual se activa este sistema. A este respecto, el modelo más estudiado es A. nidulans, en el que se encontró que dicha activación se da a través de una cascada de regulación que involucra ubiquitinación-desubiquitinación.
Por otro lado, los estudios más recientes que utilizan las nuevas tecnologías disponibles han demostrado que la función regulatoria de estos genes, ya sean reguladores positivos o negativos, es mucho más compleja de lo que se conocía, implicando la activación, la modulación y la represión de muchos otros genes, además de las celulasas, a través de redes complejas de regulación génica. Dilucidar los mecanismos de regulación de estos sistemas es fundamental, no solamente para el avance del conocimiento básico, sino desde un punto de vista biotecnológico para controlar de una manera más eficiente las condiciones tanto externas (medio ambiente) como internas (regulación de la expresión en el microorganismo), necesarias para la hidrólisis eficiente de la celulosa, de manera que esta reserva renovable de carbono pueda ser utilizada como una alternativa real, factible técnica y económicamente, a las fuentes de carbono no renovables.
Conflicto de interesesLas autoras declaran no tener ningún conflicto de intereses.
A la Universidad Nacional de Colombia y a BIOCULTIVOS S. A, por la financiación del proyecto «Desarrollo de un bioinoculante para la incorporación de residuos de cosecha mediante su descomposición in situ empleando microorganismos lignocelulolíticos», dentro del cual se enmarca este trabajo. Y a todos aquellos que contribuyeron con sus observaciones a la redacción de este manuscrito.