Dentro del género Candida, la especie más frecuentemente aislada de materiales clínicos es Candida albicans. Debido a la emergencia de otras especies que pueden presentar mayor índice de resistencia a los antifúngicos, se hace necesaria la identificación rápida de aquellas. El objetivo del presente trabajo fue evaluar el rendimiento del sistema RapID™ Yeast Plus a partir de subcultivos en 2 formulaciones diferentes, agar Sabouraud dextrosa modificado por Emmons (medio indicado en el inserto del equipo) y agar Sabouraud glucosado, que es el utilizado habitualmente en los laboratorios de la Ciudad Autónoma de Buenos Aires. Se estudiaron 166 cepas de muestras clínicas provenientes de los distintos hospitales que integran la Red de Micología de la Ciudad de Buenos Aires. De los resultados obtenidos se concluye que se deben mantener las condiciones y el medio de cultivo indicado por el fabricante.
Within the genus Candida, Candida albicans is the most commonly isolated species from clinical samples. Due to the emergence of other species which can show a higher index of antifungal resistance, a fast identification of these species is necessary. The aim of this work was to evaluate the performance of the RapID Yeast Plus system from two different subculture media formulations: Sabouraud dextrose agar adjusted by Emmons (the medium is indicated in the equipment insert) and Sabouraud glucose agar, which is the most frequently used in Buenos Aires City laboratories. One hundred and sixty-six clinical sample strains coming from different hospitals belonging to the Mycology Network of Buenos Aires City were studied. From the obtained results, we conclude that the conditions and culture medium indicated by the manufacturer should be followed.
La candidiasis invasiva es la enfermedad fúngica más común entre los pacientes hospitalizados, comprende la candidemia y la candidiasis de tejido profundo. El diagnóstico y tratamiento temprano de estas afecciones se asocia a un mejor pronóstico. Los episodios de candidemia son un problema médico creciente, sobre todo en pacientes críticos o enfermos con inmunodeficiencias o neutropenia; la candidemia representa una de las micosis oportunistas más frecuentes en todo el mundo.
La incidencia de candidemia en EE. UU. es de 0,28 a 0,96 casos por 1000 ingresos; en Europa es de 0,20 a 0,38 y en Latinoamérica es de 0,98, con una amplia variación entre los distintos países; por ejemplo, en Chile es de 0,32 por 1000 ingresos8.
En Argentina la incidencia es de 1,75 por 1000 ingresos y en la ciudad autónoma de Buenos Aires (CABA) es de 1,15 por 1000 ingresos7.
Candida albicans es el principal aislamiento clínico, pero debido a la emergencia de otras levaduras, principalmente aquellas que demuestran mayor resistencia a algunos antifúngicos, se hace necesaria la identificación rápida a nivel de especie de estos microorganismos10,11.
La utilización de medios con sustratos cromogénicos facilita la identificación presuntiva de algunas de las especies de Candida y permite reconocer la presencia simultánea de distintas levaduras en una misma muestra clínica1,2.
La identificación definitiva se realiza evaluando la asimilación y fermentación de azúcares. Para ello se encuentran disponibles métodos rápidos comerciales, como API 20C AUX® y API ID 32C®, considerado el «estándar oro»; Vitek®2 (BioMerieux, Francia), que presenta una concordancia del 98,3% respecto del «estándar oro», o el BdPhoenix (Becton Dickinson, EE. UU.).
El sistema RapID™ Yeast Plus (Remel, Reino Unido) es un micrométodo cualitativo que utiliza sustratos convencionales y cromogénicos para la identificación de hongos levaduriformes aislados de muestras clínicas4. Los estudios realizados por Wadlin et al.13 muestran que este equipo permitió identificar levaduras correctamente en un 96% (n = 201), sin necesidad de pruebas complementarias y en aproximadamente 4h. Según Sanguinetti et al. se identificaron correctamente el 95,5% (n=750)12. Otros autores, como Espinel-Ingroff et al.3, encontraron porcentajes menores en el caso de levaduras menos frecuentes.
El medio indicado en el inserto del equipo para realizar los subcultivos de los aislamientos es el agar Sabouraud dextrosa modificado por Emmons (ASDE), pero en el ámbito de la CABA la mayoría de los laboratorios de micología utilizan el agar Sabouraud glucosado (ASD), cuyo pH ácido favorece el desarrollo de los dermatofitos, aunque puede no crecer Blastomyces dermatitidis. Ambos medios se utilizan para el aislamiento y la identificación de diferentes hongos.
El objetivo del presente trabajo fue evaluar el rendimiento del sistema RapID™ Yeast Plus para la identificación de las levaduras más frecuentes en clínica médica, a partir de subcultivos en 2 formulaciones diferentes de agar Sabouraud: ASD y ASDE.
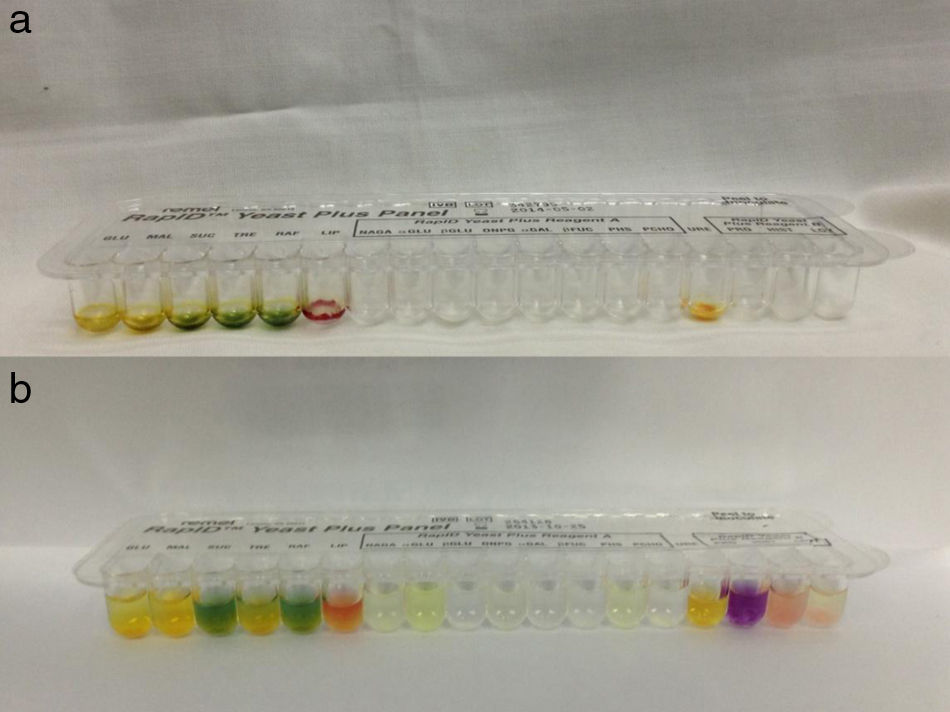
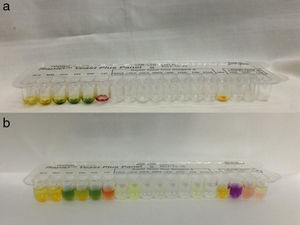
El sistema RapID™ Yeast Plus se compone de paneles RapID™ Yeast Plus, reactivo RapID™ Yeast Plus A y reactivo RapID™ Yeast Plus B. Cada panel RapID™ Yeast Plus tiene 18 pocillos moldeados en la periferia de una bandeja de plástico desechable. Los pocillos de reacción contienen los distintos sustratos deshidratados y la bandeja permite la inoculación simultánea de cada uno de ellos con una cantidad predeterminada de inóculo. Como inóculo para rehidratar e iniciar las reacciones de prueba se usa una suspensión del microorganismo que se desea identificar en el líquido de inoculación RapID™. Luego de 4h de incubación a 30°C, se examina la reactividad de cada pocillo de prueba observando el desarrollo de un color. Algunos pocillos requieren el agregado de reactivos (A o B) para obtener el cambio de color. El patrón resultante de puntuaciones positivas y negativas se usa como base para identificar el aislamiento en estudio y se comparan los resultados obtenidos con los patrones de reactividad almacenados en la base de datos o se utiliza un compendio de códigos generado por computadora. La identificación del aislamiento se expresa en porcentaje de probabilidad y el nivel de probabilidad es clasificado en: implícito, satisfactorio, adecuado, cuestionable e inaceptable. Se complementa la identificación con biopuntuación, clasificando la biofrecuencia en las siguientes categorías: típica, aceptable, rara, muy rara y no aplicable.
Las pruebas usadas en el sistema RapID™ Yeast Plus se basan en la degradación microbiana de sustratos específicos detectados por varios sistemas indicadores. Los reactivos utilizados son una combinación de pruebas convencionales y cromogénicas de monosustrato (figura 1).
Se estudiaron en total 166 cepas que pertenecieron a las siguientes especies: 39 C. tropicalis, 31C. albicans, 28 C. parapsilosis, 24 C. glabrata, 23 C. krusei y 21 C. guilliermondii. Como control se incluyeron las siguientes cepas: ATCC 22019 (C. parapsilosis), ATCC 6258 (C. krusei) y ATCC 64548 (C. albicans).
Se tomó como «estándar oro» para la identificación de las levaduras el equipo de API ID32C, más la micromorfología característica de cada levadura en agar harina de maíz6 o agar leche9 con 1% de Tween 80.
Antes de realizar el estudio, cada aislamiento se repicó en ASD a 35°C para confirmar su viabilidad. Se corroboró la pureza y se efectuó la identificación presuntiva mediante la utilización de un agar cromogénico, Brilliance Candida Agar (Oxoid, Reino Unido), y la observación de la micromorfología en el agar harina de maíz (Oxoid, Reino Unido) con Tween 80.
Una vez confirmadas ambas condiciones, se repicó cada una de las cepas en ASDE (2% de glucosa, pH 6,9) y en ASD (4% de glucosa, pH 5,6) a 30°C durante 48h. Luego de ese lapso, las 2 series de aislamientos se inocularon en los paneles RapID™ Yeast Plus.
Se evaluó el porcentaje de concordancia definido como la proporción de coincidencias entre el «estándar oro» y la identificación obtenida por el sistema RapID™ Yeast Plus.
Estudio estadístico: grado de concordancia según el índice kappa5.
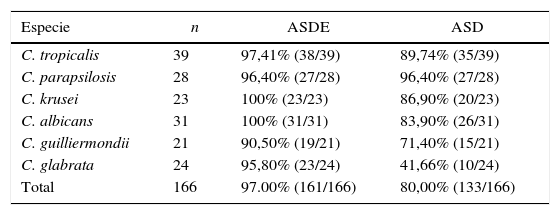
Se estudiaron 166 aislamientos y se encontró una concordancia con el «estándar oro» del 97% partiendo de ASDE (161/166) y del 80% partiendo de ASD (133/166) (tabla 1). Se observó discrepancia con respecto al «estándar oro» en 5 cepas con ASDE y en 33 cepas con ASD (tabla 2).
Grado de concordancia de resultados entre el sistema Rapid™ Yeast Plus y el método “estándar oro”
| Especie | n | ASDE | ASD |
|---|---|---|---|
| C. tropicalis | 39 | 97,41% (38/39) | 89,74% (35/39) |
| C. parapsilosis | 28 | 96,40% (27/28) | 96,40% (27/28) |
| C. krusei | 23 | 100% (23/23) | 86,90% (20/23) |
| C. albicans | 31 | 100% (31/31) | 83,90% (26/31) |
| C. guilliermondii | 21 | 90,50% (19/21) | 71,40% (15/21) |
| C. glabrata | 24 | 95,80% (23/24) | 41,66% (10/24) |
| Total | 166 | 97.00% (161/166) | 80,00% (133/166) |
ASD: medio de cultivo agar Sabouraud dextrosado; ASDE: medio de cultivo agar Sabouraud dextrosa según Emmons (medio recomendado por el fabricante del equipo comercial).
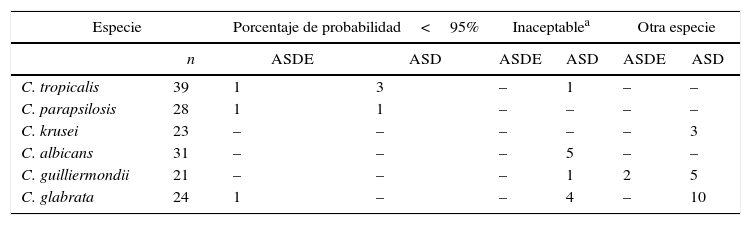
Detalle de las discrepancias de resultados entre el método «estándar oro» y el sistema RapID™ Yeast Plus
| Especie | Porcentaje de probabilidad<95% | Inaceptablea | Otra especie | ||||
|---|---|---|---|---|---|---|---|
| n | ASDE | ASD | ASDE | ASD | ASDE | ASD | |
| C. tropicalis | 39 | 1 | 3 | – | 1 | – | – |
| C. parapsilosis | 28 | 1 | 1 | – | – | – | – |
| C. krusei | 23 | – | – | – | – | – | 3 |
| C. albicans | 31 | – | – | – | 5 | – | – |
| C. guilliermondii | 21 | – | – | – | 1 | 2 | 5 |
| C. glabrata | 24 | 1 | – | – | 4 | – | 10 |
ASD: agar Sabouraud dextrosado; ASDE: medio agar Sabouraud dextrosa según Emmons (medio recomendado por el fabricante del equipo comercial).
Las aislamientos que presentaron discrepancias partiendo de ASDE correspondieron a una cepa de C. tropicalis, una cepa de C. parapsilosis y una cepa de C. glabrata con el 74,48, el 79,64 y el 81,56% de porcentaje de probabilidad, respectivamente, y 2 cepas de C. guilliermondii, que fueron identificadas como C. lusitaniae y C. lambica.
Las discrepancias partiendo de ASD se observaron en 4 aislamientos con porcentaje de probabilidad<95%, 3 aislamientos de C. tropicalis y uno de C. parapsilosis. Once aislamientos no pudieron ser identificados, ya que presentaron un nivel de probabilidad inaceptable (5 cepas de C. albicans, 4 de C. glabrata, uno de C. guilliermondii y uno de C. tropicalis). Dieciocho aislamientos fueron identificados como otra especie, 3 cepas de C. krusei fueron identificadas como: Saccharomyces cerevisiae (2) y Prototheca zopfii (1). Cinco aislamientos de C. guilliermondii fueron identificados como C. intermedia (2) y C. famata (3). Los 10 aislamientos de C. glabrata identificados como otra especie correspondieron a P. zopfii (6), S. cerevisiae (2), C. tropicalis (1) y Blastoschizomyces capitatus (1), con biofrecuencias de no aplicable, rara y muy rara.
El índice de concordancia obtenido entre ambos medios fue de 0,04, que según la escala de valoración de Landis y Koch corresponde a un grado de acuerdo insignificante (> 0,00-0,20).
De acuerdo con el grado de concordancia obtenido con el índice kappa (0,04), ambos medios no son similares, por lo que es necesario partir del ASDE.
La concordancia con el «estándar oro» varía de acuerdo con la especie de Candida y al medio de cultivo utilizado. En vista de los resultados aquí obtenidos, se concluye que se deben mantener las condiciones y el medio de cultivo indicados por el fabricante cuando se emplea el sistema Rapid™ Yeast Plus para la identificación de hongos levaduriformes aislados de muestras clínicas.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
A la Dra. Adriana Bau de la Empresa Bioartis, por la provisión de los equipos de Rapid™.
Yeast Plus (Remel), Brilliance Candida Agar, Agar Sabouraud glucosado y Agar Sabouraud dextrosa modificado por Emmons, para la realización de este trabajo en la Red de Micología- CABA.
Ivana Maldonado (Htal. Alemán), Mariela Schijman (Htal. Álvarez), Laura López Moral (Htal. Argerich), Juan Rébori (Htal. Borda), Silvia Relloso (CEMIC), Rosana Pereda (Htal. Elizalde), Analía Fernández (F. Favaloro), Mario Bianchi (Htal. Muñiz), Nora Franco (Htal. Piñero), Claudia Garbasz (Htal. Pirovano), Mónica López (Htal. Ramos Mejía).