El intestino alberga un complejo ecosistema de comunidades microbianas. Los datos experimentales sugieren que la microbiota ejerce funciones metabólicas que contribuyen a la recuperación de nutrientes y energía de sustratos no digeribles. Además, la colonización microbiana es esencial para el desarrollo normal del sistema inmune, de modo que parece influir en la homeostasis entre carga antigénica ambiental y respuesta inmune. En individuos con susceptibilidad genética, el desequilibrio puede dar lugar a patologías de disregulación inmunitaria, incluyendo las enfermedades inflamatorias crónicas del intestino, en las que el sistema inmune reacciona exageradamente frente a antígenos microbianos no nocivos. A pesar de que se dispone de nuevas tecnologías moleculares, la composición normal de la microbiota del intestino humano es todavía desconocida. Se esperan avances importantes en los próximos años por la puesta en marcha de proyectos internacionales encaminados a describir con precisión el impacto de la microbiota en diversos procesos fisiológicos y patológicos.
The intestine hosts a complex ecosystem of microbial communities. Experimental data suggests that the microbiota has metabolic functions that contribute to nutrient and energy recovery from non-digestible substrates. Moreover, microbial colonization is essential for the normal development of the immune system and therefore seems to influence homeostasis between environmental antigen load and immune response. In genetically-susceptible individuals, an imbalance may give rise to diseases of immune dysregulation, including chronic inflammatory bowel diseases, in which there is an exaggerated immune response to harmless microbial antigens. Despite the availability of new molecular technologies, the normal composition of the human intestinal microbiota remains unknown. In the next few years, the results of international projects designed to determine the precise impact of the microbiota in various physiological and pathological processes will hopefully lead to major advances.
Se han encontrado microfósiles de bacterias que los expertos datan en más de 3.500 años de antigüedad1. Poco después de que se enfriara la corteza terrestre y en zonas cubiertas por aguas poco profundas, proliferaron comunidades microbianas de cianobacterias capaces de sintetizar compuestos hidrocarbonados utilizando energía solar. Mediante un mecanismo de fotosíntesis dichas comunidades bacterianas generaron oxígeno libre que se fue acumulando en la atmósfera, y también desarrollaron vías metabólicas de consumo respiratorio de oxígeno en las fases de ausencia de luz solar. Hoy, las cianobacterias y otras comunidades microbianas siguen siendo los habitantes más importantes del planeta Tierra por su continua actividad en la conservación y equilibrio de las condiciones bioambientales: los ciclos del agua y los gases, incorporación de minerales inorgánicos a la materia orgánica, reciclado de compuestos orgánicos, etc.2.
En el siglo xvii, Anton van Leeuwenhoek describió sus observaciones del mundo microbiano usando un microscopio de lente simple diseñado por él mismo. No fue hasta mediados del siglo xix cuando la medicina entendió que las bacterias y otros micro-organismos podían ser causa de enfermedad en el hombre y los animales. La tarea investigadora de Pasteur, Koch y otros, puso en evidencia que muchas de las enfermedades de carácter transmisible, las enfermedades infecciosas, tenían un vector microbiano específico que explicaba su difusión. A lo largo de los siglos xix y xx, la medicina desarrolló métodos para evitar y combatir las enfermedades infecciosas. Las medidas de limpieza, higiene y saneamiento urbano (dotación de agua potable y sistemas de alcantarillado para recogida de aguas residuales), el desarrollo de vacunas, el descubrimiento de los antibióticos, etc., figuran entre los hitos y logros más trascendentes de la historia de la medicina3. El reconocimiento de los microbios como causa de enfermedad, junto con las medidas para prevenir y combatir infecciones, ha tenido un impacto extraordinario y sin precedentes en la sanidad pública.
Sin embargo, el homo sapiens al igual que los demás mamíferos ha vivido permanentemente en íntima asociación con diversas comunidades microbianas desde que se iniciara la existencia de la especie humana hace unos 200.000 años. La estabilidad de la asociación durante milenios refleja que existe adaptación mutua entre las distintas especies y el anfitrión, y además indica que en la relación predomina la simbiosis o mutualismo, es decir, la asociación conlleva algún tipo de beneficio para las especies implicadas incluyendo al anfitrión. Aunque la medicina se ha beneficiado mucho de reconocer y combatir el potencial nocivo de los microbios patógenos, los avances científicos subrayan que la inmensa mayoría de los microorganismos que viven en asociación con el hombre contribuyen a su buen desarrollo fisiológico.
El ecosistema intestinalEl término «microflora» o «microbiota» hace referencia a la comunidad de micro-organismos vivos reunidos en un nicho ecológico determinado. El tubo digestivo del ser humano alberga una población numerosa, diversa y dinámica de microorganismos, principalmente bacterias, pero también hongos, virus y eucarias (o protozoos), que se han adaptado a la vida en las superficies mucosas o en la luz del intestino desde hace milenios4. Buena parte de los micro-organismos que habitan el intestino humano se llaman nativos o indígenas porque habitan permanentemente el tracto gastrointestinal, pero sería erróneo interpretar el apelativo de ‘nativos’ como indicativo de microorganismos autogenerados in situ. De hecho, el individuo nace completamente estéril y adquiere su colonización microbiana inmediatamente después del nacimiento, fundamentalmente de la madre y del entorno ambiental inicial. Otras bacterias son microorganismos en tránsito, porque habitan temporalmente la luz o las mucosas y después desaparecen. Estas bacterias se ingieren continuamente del ambiente (fundamentalmente a través de comidas y bebidas, etc.). El conjunto conforma el ecosistema microbiano del intestino, una colectividad de seres vivos con interdependencia y mutualismo entre las especies que lo constituyen.
Se estima que la población microbiana del intestino humano incluye unos 100 billones de bacterias de unas 500 a 1.000 especies distintas, de modo que el número de células bacterianas es 10 veces más grande que el número de células somáticas. El estómago y el duodeno albergan un reducido número de microorganismos que se adhieren a la superficie mucosa o están en tránsito, habitualmente menos de 103 células bacterianas por gramo de contenido. Las secreciones ácidas, biliares y pancreáticas destruyen la mayor parte de los microorganismos ingeridos, y la actividad motora propulsiva impide una colonización estable de la luz del intestino delgado. El número de bacterias aumenta progresivamente a lo largo del yeyuno y el íleon, desde alrededor de 104 en el yeyuno hasta 107 unidades formadoras de colonias por gramo de contenido en el extremo ileal, con un predominio de aerobios gramnegativos y algunos anaerobios obligados. En el colon el tiempo de tránsito es lento (2-4 días) lo que brinda a los microorganismos la oportunidad de proliferar fermentando los sustratos disponibles derivados de la dieta o de las secreciones endógenas. El colon está densamente poblado de anaerobios y los recuentos de bacterias alcanzan densidades de alrededor de 1011 unidades formadoras de colonias por gramo de contenido (concentraciones 10.000 veces mayores que en la luz ileal). El intestino humano junto con el de otros vertebrados es uno de los ecosistemas más densamente poblados de la tierra.
En años recientes se han adquirido suficientes conocimientos para poder afirmar que las funciones del tubo digestivo, nutrición y defensa, dependen no solo de las estructuras propias (barrera mucosa, glándulas secretoras, sistema inmune de las mucosas) sino también de la presencia y actividad de las comunidades microbianas que colonizan el intestino. La microbiota intestinal es un órgano más, perfectamente integrado en la fisiología del individuo4. Tubo digestivo y microbiota son interdependientes y hay evidencias que sugieren que su equilibrio condiciona la homeostasis del individuo dentro de su entorno ambiental.
Las evidencias más aplastantes se obtuvieron experimentalmente en animales criados en condiciones de asepsia total (germ-free)5. En estos animales, el peso total corporal y el de los órganos vitales (corazón, hígado, pulmones) es menor que en los controles criados de modo convencional (SPF, specific pathogen free, en contacto con bacterias pero sin patógenos). Sin embargo los germ-free ingieren mayores cantidades de alimento, es decir, necesitan comer más pero crecen menos. Esto indica claramente que la presencia de bacterias es importante para el rendimiento nutricional y el desarrollo corporal. Por otra parte, los animales sin gérmenes presentan ganglios linfáticos atróficos, tienen menos linfocitos en la mucosa intestinal y niveles bajos de inmunoglobulinas plasmáticas. En estas condiciones, los animales germ-free muestran mayor susceptibilidad a infecciones, lo que significa que la presencia de bacterias es necesaria para que el sistema inmune se desarrolle adecuadamente.
Funciones de la microbiotaA partir de datos experimentales obtenidos mediante estudios en animales con colonización intestinal controlada, se han identificado tres funciones primarias de la microbiota intestinal6: a) funciones de nutrición y metabolismo, como resultado de la actividad bioquímica de la microbiota, que incluyen recuperación de energía en forma de ácidos grasos de cadena corta, producción de vitaminas y efectos favorables sobre la absorción de calcio y hierro en el colon; b) funciones de protección, previniendo la invasión de agentes infecciosos o el sobrecrecimiento de especies residentes con potencial patógeno, y c) funciones tróficas sobre la proliferación y diferenciación del epitelio intestinal, y sobre el desarrollo y modulación del sistema inmune.
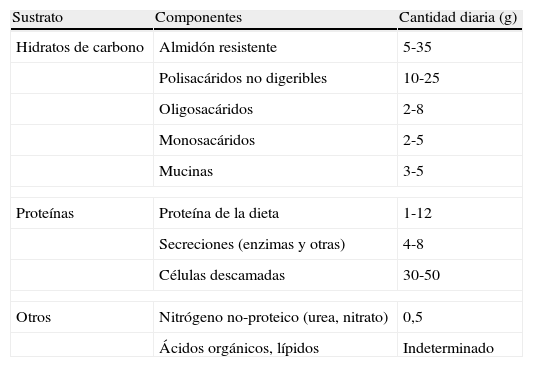
La microbiota como órgano metabólicoLa microbiota residente en el colon metaboliza los sustratos o residuos dietéticos no digeribles, el moco endógeno y los detritus celulares. La tabla 1 muestra información sobre sustratos que alcanzarían el ciego en pacientes portadores de ileostomía quirúrgica7. La fermentación de polisacáridos vegetales de estructura compleja (fibras) tiene lugar fundamentalmente en ciego y colon derecho. Constituye una fuente de energía importante para la proliferación bacteriana, y además produce ácidos grasos de cadena corta que el anfitrión puede absorber. Esto se traduce en recuperación de energía de la dieta y también favorece la absorción de iones (Ca, Mg, Fe) en el ciego.
Sustratos metabolizables en el colon humano
| Sustrato | Componentes | Cantidad diaria (g) |
| Hidratos de carbono | Almidón resistente | 5-35 |
| Polisacáridos no digeribles | 10-25 | |
| Oligosacáridos | 2-8 | |
| Monosacáridos | 2-5 | |
| Mucinas | 3-5 | |
| Proteínas | Proteína de la dieta | 1-12 |
| Secreciones (enzimas y otras) | 4-8 | |
| Células descamadas | 30-50 | |
| Otros | Nitrógeno no-proteico (urea, nitrato) | 0,5 |
| Ácidos orgánicos, lípidos | Indeterminado | |
Datos obtenidos de pacientes con ileostomía.
Fuente: Egert M, et al7.
La fermentación microbiana de polisacáridos complejos, celulosas y ligninas, es la fuente principal de energía de la que depende el desarrollo vital de los mamíferos herbívoros8. No era muy distinta la situación de dependencia energética en el caso de nuestros antepasados humanos, antes de que se dispusiera de recursos agrícolas o ganaderos estables. Algunos estudios arqueológicos estiman que la cantidad de sustratos vegetales no digeribles que alcanzaba el colon en nuestros antecesores de hace 10.000 años alcanzaba los 250 gramos por día, mucho mayor que en la actualidad.
Experimentos recientes en ratones criados en condiciones germ-free demuestran cómo el acúmulo de energía en forma de tejido adiposo depende de la microbiota residente en el intestino8. Por un lado, la microbiota genera energía absorbible a partir de polisacáridos complejos. Pero además, el trofismo de las bacterias sobre el epitelio intestinal tiene efectos a distancia, facilitando el depósito de triglicéridos en el tejido adiposo, mediante la regulación de la producción epitelial de un factor humoral8. Las funciones metabólicas también incluyen la producción de vitaminas (K, B12, biotina, ácido fólico y pantoténico) y la síntesis de aminoácidos a partir del amoniaco o la urea. El metabolismo anaeróbico de los péptidos y proteínas (putrefacción) tiene lugar en los tramos más distales del intestino grueso5. La putrefacción produce ácidos grasos de cadena corta, pero, al mismo tiempo, genera una serie de sustancias potencialmente tóxicas incluido el amoniaco, aminas, fenoles, tioles e indoles.
Homeostasis del sistema inmuneLa presencia de microbiota en el intestino tiene un impacto decisivo sobre el desarrollo y maduración del sistema inmune9. La concepción de que el sistema inmune se desarrolla en respuesta a estímulos infecciosos provocados por microorganismos patógenos no parece sostenerse. Se ha demostrado que la colonización intestinal permanente por bacterias comensales, no necesariamente patógenas, es el factor esencial. Los animales criados en condiciones de asepsia estricta muestran atrofia del sistema inmune de las mucosas (folículos linfoides de tamaño pequeño, escasez de linfocitos intraepiteliales y de la lamina propia) y también del sistema inmune sistémico (ganglios linfáticos de menor tamaño, bajo nivel de inmunoglobulinas). La colonización bacteriana incrementa rápidamente el número de linfocitos de la mucosa, los centros germinales de los folículos linfoides y ganglios crecen en número y tamaño, aparecen células productoras de inmunoglobulinas en la lámina propia, y los niveles de inmunoglobulinas séricas se normalizan10. La inducción y regulación del sistema inmune depende de la colonización bacteriana del tracto digestivo.
La superficie de la mucosa del tracto digestivo es un área dotada de estructuras adaptadas a funciones de contacto y comunicación entre el individuo y el medio externo. Esta comunicación incluye el reconocimiento inmunológico de las sustancias del exterior que transitan por el tubo digestivo9. Para la perfecta homeostasis, el sistema tiene que distinguir claramente entre patógenos o patógenos potenciales, de un lado, y microbios comensales en simbiosis con el anfitrión, de otro. En el primer caso, el organismo debe dotarse de elementos de defensa adecuados, mientras que en el segundo caso, el anfitrión tiene que saber tolerar para obtener el beneficio de la simbiosis. Las interacciones entre los microorganismos, el epitelio y los tejidos linfoides intestinales son múltiples, diversas en sus características y continuas, de modo que remodelan constantemente los mecanismos locales y sistémicos de la inmunidad adaptándolos al ambiente microbiano11.
Los enterocitos del epitelio de la mucosa intestinal tienen un papel muy importante en la logística del sistema inmune. Su posición en primera línea y en contacto con la luz intestinal es crucial para el reconocimiento inicial de moléculas foráneas y para la generación de señales que se transmiten a las células inmunocompetentes del tejido subyacente. La activación de los mecanismos de defensa depende en primer lugar del reconocimiento rápido de riesgo a través de receptores innatos o pre-formados que detectan componentes estructurales comunes a bacterias o virus12. Esto se realiza en el medio extracelular mediante los Toll-like-receptors (TLR) de la membrana, y en el medio intracelular mediante las proteínas tipo NOD del citosol. La activación de estos sensores por invasión bacteriana genera inmediatamente señales que convergen en la migración de factores de transcripción (NF-kappaB y otros) al núcleo celular, donde activan la expresión de genes responsables de la síntesis de proteínas proinflamatorias, básicamente citoquinas y enzimas inducibles con capacidad para generar mediadores inflamatorios. De este modo, las células epiteliales emiten señales con capacidad de atraer y activar leucocitos, aumentar el flujo sanguíneo, incrementar la permeabilidad capilar, etc. Los enterocitos pueden actuar como células presentadoras de antígenos, sugiriendo que su rol no se limita a la defensa innata sino que también participan en el escalón inicial de las respuestas de tipo adquirido (expansión de clones linfocitarios específicos y generación de anticuerpos).
La mucosa intestinal cuenta con elementos y estructuras de tejido linfoide inmuno-competente con funciones especializadas de defensa adquirida9. El llamado ‘sistema inmune de las mucosas’ incluye tres compartimentos diferenciables anatómicamente: estructuras organizadas (placas de Peyer y folículos linfoides), lámina propia y epitelio superficial. Las estructuras organizadas son lugares de inducción, mientras que la lámina propia y el compartimiento epitelial contienen células maduras y efectoras. Las estructuras organizadas están cubiertas por epitelio especializado (células M, de morfología característica), que transporta microorganismos o estructuras antigénicas desde la luz hasta el tejido linfoide subyacente. La inducción de respuestas inmunes de tipo adquirido es un fenómeno que tiene lugar principalmente en las estructuras foliculares de la mucosa intestinal. Los antígenos procesados por células presentadoras de antígenos (por ejemplo, células dendríticas) se presentan a linfocitos T en estado naïve, y se activa la expansión de los clones más afines al antígeno. La expansión clonal de células T da lugar a linfocitos helper (células Th) de distinto fenotipo: Th1, Th2 o T reguladoras (Th3, Tr1 o células CD4-CD25). Las células T reguladores desempeñan un papel central en inmunotolerancia porque segregan citocinas reguladoras, de carácter antiinflamatorio (IL-10, TGF-beta), en respuesta a antígenos que se reconocen como no patógenos. En condiciones normales, predomina un contexto de inmunotolerancia probablemente mediado por células T reguladoras. Este contexto de inmunotolerancia permite la exposición continua a una carga antigénica abrumadora (microbiota, comida, antígenos ambientales), sin que por ello se desencadenen reacciones inflamatorias que lesionarían al tejido intestinal propio11,13,14.
Composición de la microbiota intestinalHasta hace menos de una década, nuestros conocimientos sobre la composición bacteriana de la microbiota intestinal se basaban principalmente en la información obtenida por cultivo de muestras de heces o de biopsias intestinales6. El análisis bacteriológico convencional de la flora fecal por aislamiento de bacterias en medios de crecimiento selectivo demuestra que las bacterias anaerobias estrictas superan en número a las aerobias por un factor de al menos 100 o 1.000 especies anaerobias por cada especie aerobia. Según la metodología tradicional, los géneros predominantes son Bacteroides, Bifidobacterium, Eubacterium, Clostridium, Lactobacillus, Fusobacterium y diversos cocos anaerobios. No obstante, los investigadores siempre han sido conscientes de que la información obtenido por cultivo es incompleta, en primer lugar porque las técnicas de cultivo de anaerobios en el laboratorio tienen muchas limitaciones, y en segundo lugar, porque más del 50% de las células bacterianas observadas mediante examen microscópico de muestras fecales no se recuperan en medios de cultivo6.
A lo largo de las últimas dos décadas se han introducido diversas técnicas de biología molecular para identificar y caracterizar las bacterias no cultivables15. Las nuevas tecnologías están proporcionando información completamente novedosa sobre el ecosistema del intestino. Por medios electroforéticos se pueden separar el ADN bacteriano en bandas, y cada una de ellas contiene grupos bacterianos con secuencias de ADN parecidas. Este tipo de estudios refleja que cada individuo tiene un patrón de bandas muy característico y estable a lo largo del tiempo. Parece que el patrón o perfil viene determinado en buena parte por el genotipo del propio individuo que selecciona su propia microbiota, en tanto que es más parecido entre hermanos gemelos, aunque ya no convivan, que entre parejas estables que conviven, compartiendo alimentos y alojamiento16. Sin embargo, también se detecta claramente la influencia del ambiente, en tanto que los individuos hospitalizados en una misma institución, pierden diversidad, especialmente si están sometidos a tratamiento antibiótico17. Otro estudio hospitalario basado en la misma técnica de bandas de ADN ha demostrado que los recién nacidos, que están en su periodo de adquisición de la microbiota, son especialmente sensibles a la influencia del ambiente, ya que presentan una serie de bandas comunes tras su estancia en la unidad de neonatos18.
Algunas de estas técnicas utilizan la diversidad de secuencias de un gen bacteriano, el 16S de ARN ribosomal. Se trata de un gen altamente conservado (todas las bacterias tienen ribosomas) y su secuenciación ha permitido la realización de estudios filogenéticos y taxonómicos (genealogía de géneros y especies). Se dispone de un gran número de secuencias almacenadas en el GenBank de modo que la secuenciación de fragmentos del gen 16S rRNA permite la identificación o al menos la clasificación taxonómica de las bacterias presentes en una muestra15. Cabe destacar que los estudios iniciales con esta técnica sirvieron para detectar que cada individuo alberga un gran número de cepas propias o únicas, es decir que no se encuentran en otros individuos, y que además no se encontraban en las bases de datos. Gracias a esta tecnología se han descubierto nuevos géneros bacterianos que habían pasado totalmente desapercibidos a pesar de su gran abundancia y presencia en el intestino humano, por ser microorganismos de difícil aislamiento en cultivo de laboratorio. Por otro lado, estas técnicas muestran diferencias en las especies predominantes entre el tercio proximal y distal del colon19, y entre las comunidades mucosa y fecal20.
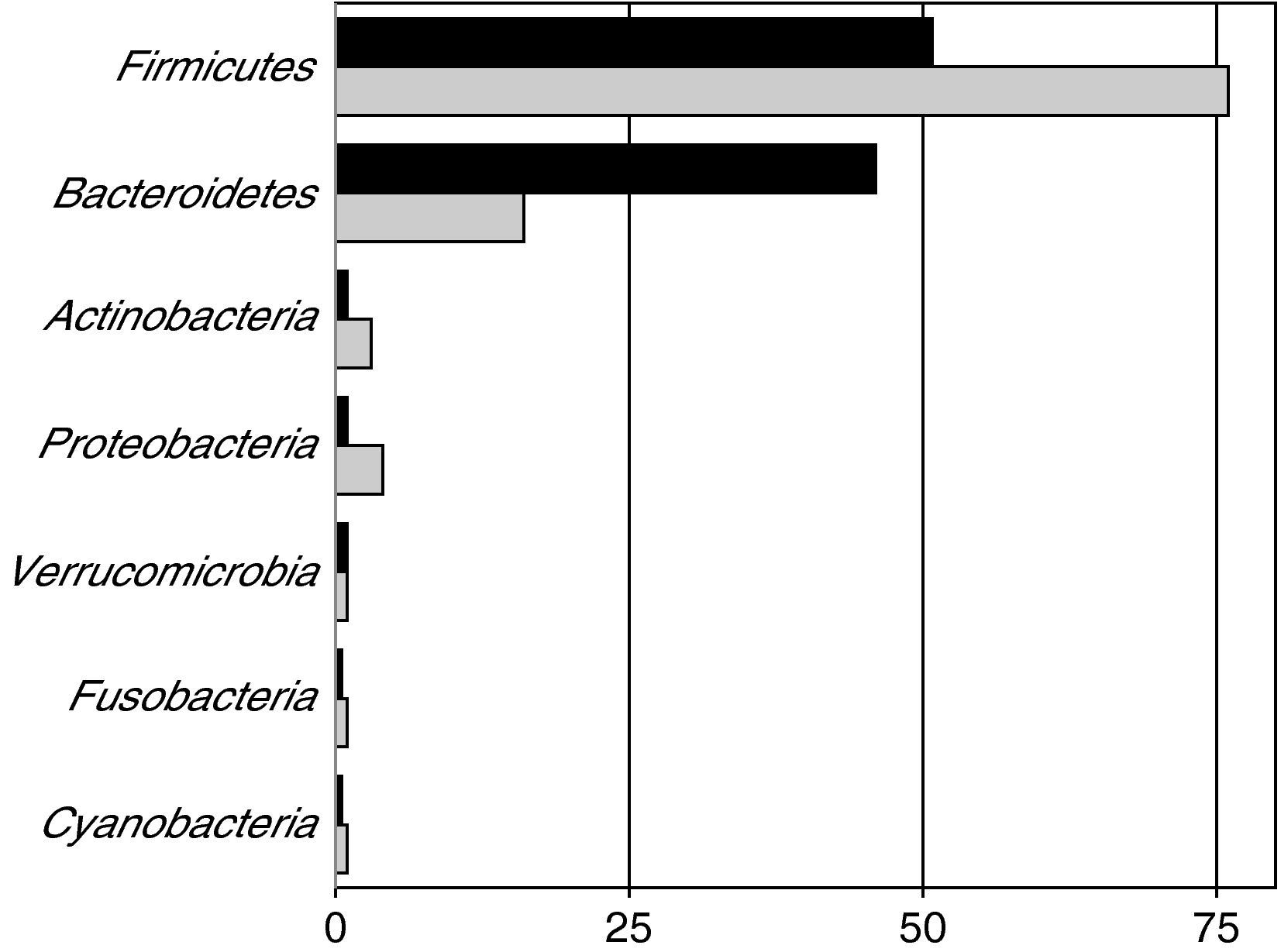
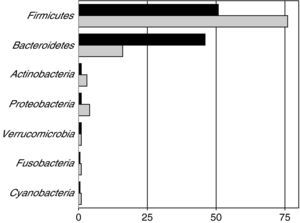
El gen 16S rRNA y metagenómicaLa metodología molecular basada en secuenciación del gen 16S rRNA ofrece una visión mucho más completa del ecosistema microbiano del intestino humano21. En muestras de mucosa intestinal y de la luz intestinal se reconocen secuencias del 16S rRNA pertenecientes a 8 clases o divisiones del dominio biológico “Bacteria”, aunque el 90% de las secuencias pertenecen a tres clases principales: Firmicutes, Bacteroidetes o Actinobacteria (fig. 1). El dominio ‘Bacteria’ está constituido por 55 clases, y cabe mencionar que otros ecosistemas conocidos incluyen un número mayor de divisiones, por ejemplo, en el suelo se encuentran especies de 20 clases de bacterias, y lo mismo ocurre en reservorios naturales de agua. Sin embargo, las 8 clases bacterianas presentes en el intestino humano están representadas con gran diversidad, de modo que hay una inmensa variedad de géneros, especies y cepas dentro de cada clase. Así, se observa que hay poca comunidad de cepas entre distintos individuos. Los estudios moleculares a nivel de especie o de cepa sugieren que cada individuo tiene una composición bacteriana muy ‘personalizada’ con relativamente pocos rasgos comunes entre distintos individuos. Por ejemplo, un análisis exhaustivo de muestras de 3 individuos obtuvo 13.335 cepas distintas, y 7.555 (57%) estaban presentes solo en uno de los tres individuos21. En cambio, a nivel de clase todos los individuos humanos estudiados encajan en una distribución composicional relativamente similar.
Composición bacteriana de la microbiota humana según técnicas de secuenciación del 16S rRNA.
Fuente: Eckburg PB, et al21.
La secuenciación del gen 16S rRNA permite la identificación taxonómica de bacterias, a nivel de clase, género, especie y cepa, de modo que se puede definir la composición de un ecosistema en cuanto a su diversidad, es decir según el catalogo censal de las bacterias que están presentes. Sin embargo la lista taxonómica de composición bacteriana puede tener muy poco significado desde un punto de vista funcional, ya que las bacterias comparten un gran número de genes y funciones, aunque sean de distinto género o especie. En la actualidad los proyectos internacionales que estudian la composición de la microbiota intestinal humana se plantean como objetivo la secuenciación masiva de todos los genes presentes en una muestra (metagenómica). Los avances en tecnología de secuenciación junto a la disponibilidad de instrumentos bio-informáticos que permiten identificar y clasificar funcionalmente las secuencias están proporcionando información muy novedosa e interesante22,23.
El estudio europeo MetaHIT ha definido por primera vez el catálogo completo o casi completo de los genes microbianos que componen el metagenoma humano. El proyecto ha identificado en muestras de heces humanas más de tres millones de genes microbianos, bacterianos en un porcentaje superior al 95%, o de origen viral o eucariota (protozoos, levaduras) en el porcentaje restante22. Es interesante el dato de que en cada muestra individual se reconocen unos 600 mil genes microbianos como promedio (tabla 2). La mitad de ellos son genes relativamente comunes a la mayoría de los individuos. El estudio bio-informático del metagenoma ha identificado más de 20.000 funciones biológicas asociadas al catálogo completo de más de 3 millones de genes, y es interesante resaltar que hay unas 6.000 funciones comunes a todos los individuos. En definitiva, la composición bacteriana de la microbiota del intestino humano y su dinamismo funcional concreto todavía hoy encierran muchos enigmas, pero esperamos que los instrumentos moleculares permitan avances importantes en los próximos años.
El metagenoma humano
| Genes no humanos en el intestino | Número |
| Catálogo de genes no redundantes | 3,299,822 |
| Media de genes por individuo | 590,384 |
| Genes comunes (>50% de individuos) | 294,110 |
| Genes raros (<20% de individuos) | 2,375,655 |
Datos del proyecto MetaHIT.
Fuente: Qin J, et al22.
Tal como se ha señalado, la interacción entre las estructuras del sistema inmune de la mucosa intestinal y el mundo microbiano de la luz intestinal parece ser un mecanismo primario en la conformación del estado de inmunotolerancia activa mediado por células T reguladoras11–14. Algunas anomalías en el desarrollo del sistema inmune podrían deberse a defectos o instrucción inadecuada de las vías reguladoras. De acuerdo con la hipótesis de la higiene, en las sociedades occidentalizadas la incidencia cada vez mayor de atopias (eczema, asma, rinitis, alergias), enfermedad inflamatoria intestinal y trastornos autoinmunes (esclerosis múltiple, diabetes tipo 1) podría explicarse por una disminución de la carga microbiana en los primeros meses de vida. Hay evidencias que sugieren que la exposición a microorganismos no patógenos, incluyendo helmintos, transmitidos por los alimentos y por vía orofecal ejerce un impacto homeostático probablemente por inducción de mecanismos de tolerancia mediados por células T reguladoras. Enfermedades por disregulación inmunitaria como las alergias, esclerosis múltiple, diabetes tipo I, enfermedad de Crohn y colitis ulcerosa, se caracterizan por defectos en la actividad de la células T reguladoras24–27.
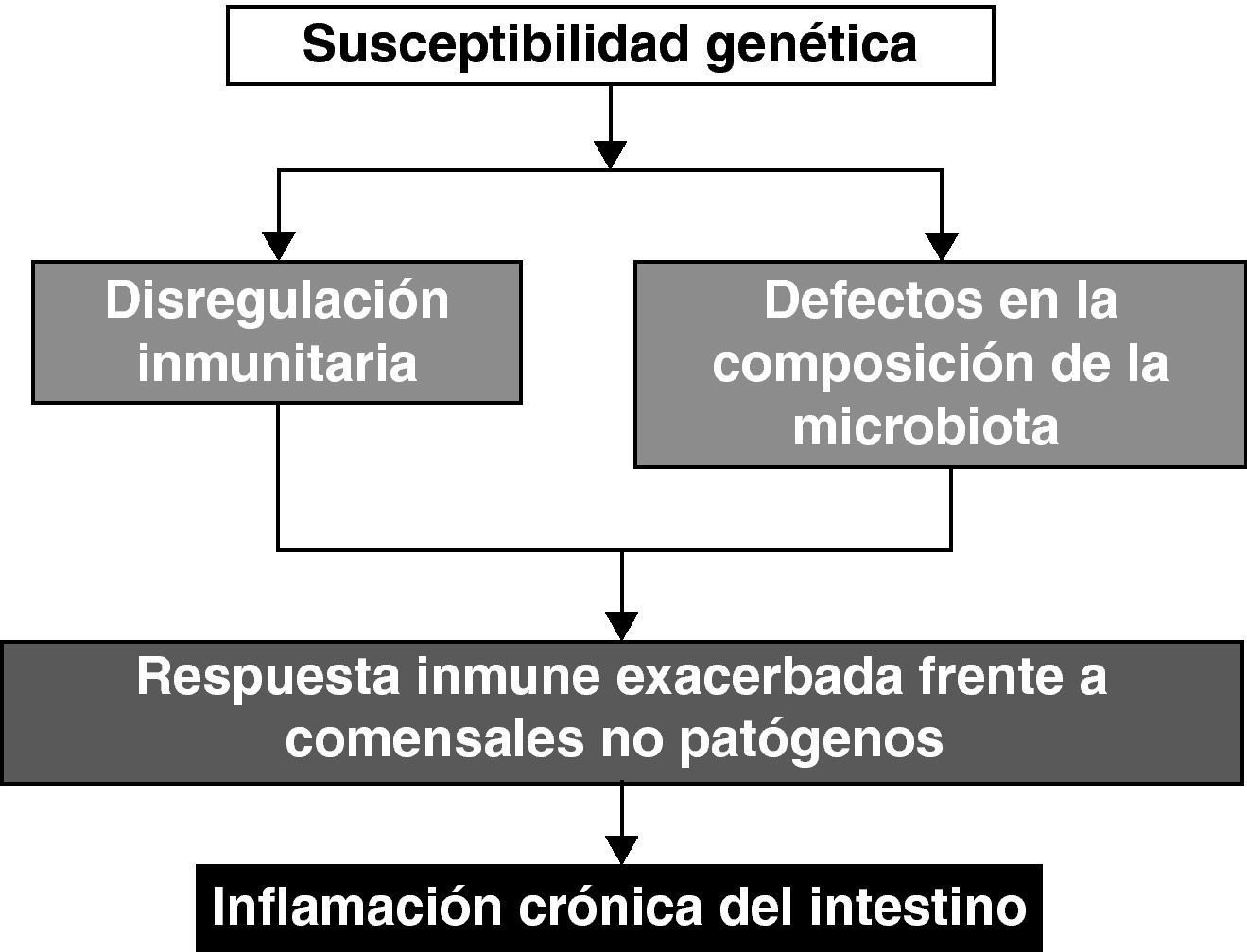
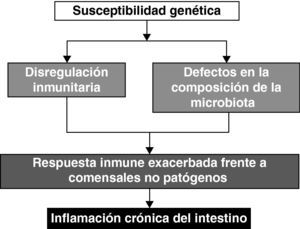
En la enfermedad de Crohn y la colitis ulcerosa existe una activación anómala del sistema inmunitario de la mucosa frente a elementos de la microbiota entérica. Esta respuesta aberrante parece ser el acontecimiento clave que desencadena los mecanismos inflamatorios que dan lugar a la lesión intestinal28. En los pacientes se detecta un aumento de la secreción mucosa de anticuerpos IgG contra las bacterias comensales29 y los linfocitos T de la mucosa son hiperreactivos frente a los antígenos de la flora común, lo que sugiere la abolición de los mecanismos de tolerancia local30. De hecho, en pacientes con enfermedad de Crohn la derivación del flujo fecal consigue remisión de las lesiones, mientras que la re-infusión del contenido intestinal en los segmentos ileales excluidos reactiva la enfermedad31. En la colitis ulcerosa, el tratamiento de la luz de colon con antibióticos de amplio espectro (administrados con recubrimiento entérico para evitar su absorción) reduce rápidamente la actividad inflamatoria32. La presencia de bacterias intestinales es por tanto un elemento clave en la patogenia de las lesiones inflamatorias. Se especula que la falta de tolerancia hacia los elementos de la propia flora sería debida a desequilibrios en su composición. La susceptibilidad genética33 y los defectos del ecosistema microbiano intestinal serían los factores condicionantes para desarrollar enfermedad inflamatoria intestinal (fig. 2).
La reactividad anormal del sistema inmune frente a las bacterias del intestino puede explicarse por la confluencia de susceptibilidad genética (NOD2, IBD15, etc.) con factores ambientales (sociedades ‘occidentalizadas’ en su desarrollo económico-social, higiene, antiinflamatorios no esteroideos, etc). Estos factores pueden ser causa de defectos en la regulación del sistema inmune y en la colonización del intestino que condicionrían una respuesta inmune inapropiada frente a elementos no nocivos de la flora.
Hay abundante información que indica que en pacientes con enfermedad de Crohn o colitis ulcerosa la población de bacterias intestinales difiere de la de los individuos sanos34. Los trabajos basados en tecnologías moleculares permiten cierta visión global del ecosistema intestinal. Algunos estudios han observado que una proporción alta de bacterias presentes en muestras fecales de pacientes con enfermedad inflamatoria pertenecen a grupos filogenéticos poco comunes en individuos sanos35,36. Esta proporción alcanza entre el 30 y 40% de las bacterias dominantes, y por tanto resulta muy significativa, pero no queda claro si es un fenómeno primario o secundario a la inflamación, aunque la anomalía no se observa en pacientes con colitis aguda de etiología infecciosa. Otros estudios demuestran que los pacientes con enfermedad inflamatoria tienen menos variedad o diversidad de especies que los individuos controles, tanto en muestras fecales como en la comunidad de bacterias asociadas a la mucosa37–40. Se ha descrito que el defecto de un organismo comensal habitual, Faecalibacterium prausnitzii, se asocia con recurrencia precoz de la enfermedad de Crohn tras cirugía ‘curativa’41. Es interesante mencionar que la baja diversidad de especies comensales se relaciona con inestabilidad en el ecosistema, que tiene mayor susceptibilidad para cambiar su composición por influencias ambientales42. La inestabilidad del ecosistema podría favorecer desequilibrios o alteraciones temporales relacionadas con riesgo de inflamación.
Otros estudios de interés han identificado un incremento de la población bacteriana adherente a la mucosa intestinal, con predominio de enterobacterias y bacteroides43. El fenómeno se relaciona con la actividad de la enfermedad, de modo que en áreas de gran inflamación se observa no solo incremento de la población adherente sino también invasión del epitelio por bacterias habituales de la flora fecal.
ConclusionesEl intestino humano alberga una comunidad diversa de bacterias comensales, en una relación de simbiosis con el individuo anfitrión, de modo que influye permanentemente en su fisiología. Hay evidencia clara de que las interacciones bacteria-anfitrión en la mucosa del intestino desempeñan un papel muy importante en el desarrollo y regulación del sistema inmune. Si esta interacción no es adecuada, la homeostasis entre la carga antigénica ambiental y la respuesta del individuo puede fallar. Ello puede repercutir en el desarrollo de patologías de disregulación inmunitaria frente a estructuras antigénicas propias, incluyendo la propia microbiota como parece ocurrir en las enfermedades inflamatorias crónicas del intestino.
En la enfermedad de Crohn y en la colitis ulcerosa, las bacterias comensales del tracto entérico constituyen la diana a la que se enfrenta el sistema inmune y por tanto el estímulo que desencadena y perpetúa los fenómenos inflamatorios que destruyen la mucosa. Muchos datos sugieren que la población bacteriana de estos individuos tiene rasgos anómalos en cuanto a su composición: grupos bacterianos poco habituales, baja diversidad y alta inestabilidad del ecosistema.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.