Escherichia coli es el principal agente causal de infecciones del tracto urinario (ITU), y sus factores de virulencia son los responsables de la gravedad de estas infecciones emergentes. El objetivo de este estudio fue evaluar la relación entre los determinantes de virulencia y susceptibilidad a antibióticos con los grupos filogenéticos de E.coli aisladas de ITU en 2 localidades de México.
MétodosSe analizaron 50 aislamientos de E.coli de una localidad en el centro del país y 57 provenientes de una localidad al suroeste. Los aislamientos fueron caracterizados fenotípica (serotipificación, ensayos de adherencia, formación de biopelícula, producción de hemolisina y susceptibilidad antibióticos) y genotípicamente (grupos filogenéticos y genes de virulencia).
ResultadosLos grupos filogenéticos B2 (60%) y F (12%) fueron significativamente predominantes en la localidad del centro con mayor frecuencia de los genes fimH (96%), iutA (66%) y sat (36%) en comparación con la localidad en el suroeste, donde los grupos A (35%) y B1 (21%) fueron más frecuentes y presentaron menor cantidad de genes de virulencia. El 21,5% del total de aislamientos pertenecieron al grupo O25-ST131. La producción de hemolisina y biopelícula fue significativamente mayor en cepas de la localidad del sureste. La resistencia a ampicilina (92,5%), tetraciclina (76,6%) y trimetoprim/sulfametoxazol (70,1%) fueron las más comunes en ambos grupos.
ConclusiónEl grupo filogenético, los factores de virulencia y la susceptibilidad a antibióticas de E.coli causante de ITU en la comunidad varían significativamente entre las poblaciones mexicanas estudiadas. Los grupos filogenéticos A y B1 pueden ser multirresistentes y tienen la capacidad de producir infecciones urinarias.
Escherichia coli is the major causative agent of urinary tract infections (UTI), and virulence factors are responsible for the severity of these emerging infections. The aim of this study was to evaluate the relationship between virulence determinants and antibiotic susceptibility with phylogenetic groups of E.coli isolates of UTI in two locations in Mexico.
MethodsAn analysis was performed on 50 isolates of E.coli from the centre of the country and 57 from a town in the southwest. The isolates were characterized by phenotype (serotyping assays, in vitro adhesion, biofilm formation, production of haemolysin, and antibiotic susceptibility) and genotype (phylogenetic groups and virulence genes).
ResultsIn the centre of the country location the phylogenetic group B2 (60%) and F (12%) were significantly more prevalent and had a higher frequency of genes, fimH (96%), iutA (66%), sat (36%), compared to the southwest location, where the group A (35%) and B1 (21%) were significantly predominant and had fewer virulence genes. About one-fifth (21.5%) of all isolates belonged to the O25-ST131 group. Haemolysin and biofilm producing strains were significantly higher in the southwest location. Resistance to ampicillin (92.5%), tetracycline (76.6%), and trimethoprim/sulfamethoxazole (70.1%) were the most common in both groups.
ConclusionThe phylogenetic group, virulence factors, and antibiotic susceptibility of the E.coli that causes UTI in the community, varies significantly among the Mexican populations studied. Phylogenetic groups A and B1 may be multidrug resistant and have the ability to produce UTI.
Escherichia coli es el agente causal más común de infecciones en el tracto urinario (ITU), incluyendo cistitis aguda, pielonefritis y urosepsis, los 3 síndromes más comunes y clínicamente distintos. Normalmente, E.coli establece una relación simbiótica con su huésped y tiene una función importante en la promoción de la estabilidad de la microbiota normal intestinal1. Sin embargo, las infecciones causadas por E.coli extraintestinales son la principal causa de morbilidad, mortalidad y altos costos asociados a la salud. Las E.coli tienen que adaptarse al medio ambiente del hospedero (vejiga, riñón y torrente sanguíneo) y los factores de virulencia juegan un papel importante en las etapas iniciales de interacción con el hospedero2. Los factores de virulencia son principalmente de 2 tipos: los expresados en la superficie celular (que desempeñan funciones de adhesión e invasión de tejidos además de formación de biopelículas e inducción de citosinas) y los producidos dentro de la célula bacteriana y que son exportados al sitio de infección3.
Los principales facilitadores de la invasión a células epiteliales de la vejiga son los pili tipo1, los cuales se expresan en más de un 90% en todos los aislamientos de E.coli, incluyendo tanto cepas patógenas como comensales. Los pili tipo1 están compuestos por repeticiones de subunidades de pilina FimA, la parte distal del pili está formada por 2 adaptadores (proteínas FimF y FimG) y la adhesina unida a manosa FimH. Esta adhesina FimH media la adherencia de la bacteria a una serie de glucoproteínas y epítopes peptídicos no glucosilados en el epitelio de la vejiga que conducen a la internalización de la bacteria, formando comunidades bacterianas intracelulares4-6. Mientras que el pili P (pili asociado a pielonefritis) se ha encontrado aproximadamente en el 80% de los aislamientos causantes de ITU altas7, las distintas subunidades estructurales de las fimbriasP se encuentran codificadas por el operón pap8.
Tres tipos de toxinas son producidas por E.coli uropatógenas (UPEC), la α-hemolisina, el factor citotóxico necrosante tipo1 (CNF1) y la toxina secretada autotransportadora Sat. La α-hemolisina (HlyA), también denominada «toxina formadora de poros», se inserta dentro de la membrana celular del huésped provocando lisis celular, facilitando la liberación de hierro y nutrientes que son esenciales para el crecimiento bacteriano9. El CNF1 conduce a una activación constitutiva de los miembros de la familia Rho, resultando en el rearreglo del citoesqueleto de la célula huésped, provocando apoptosis de las células de la vejiga estimulando su exfoliación in vivo10. La toxina Sat es una serina-proteasa que se encuentra clasificada dentro de la familia de las serina-proteasa autotransportadora de Enterobacteriaceae (SPATE), la cual se encuentra predominantemente en cepas de UPEC y se caracteriza por tener efectos citopáticos en riñón y vejiga; esta toxina puede inducir vacuolización dentro del citoplasma de células uroepiteliales11.
En función de las relaciones de similitudes evaluadas mediantes técnicas de electroforesis de diferentes enzimas y de secuenciación de sus genes (MLST) se han determinado grupos filogenéticos12. Clermont et al.13 han desarrollado una PCR cuádruple que reconoce 7 grupos filogenéticos (A, B1, B2, C, D, E, F), uno llamado clado1. Además, hay evidencias de que ciertos serotipos E.coli se asocian con ITU: O1, O2, O4, O6, O7, O8, O16, O18, O25 y O7514,15.
El manejo clínico de las ITU es complicado debido al incremento en la incidencia de infecciones causadas por cepas de E.coli que son resistentes a los antibióticos comúnmente utilizados y son productores de biopelículas. Recientemente, una clona de E.coli, O25-ST131, productora de betalactamasas de espectro extendido, multirresistentes y con alta virulencia, ha emergido a nivel mundial como una causa importante de las ITU adquiridas en la comunidad16.
A pesar de la identificación de múltiples genes asociados a la virulencia en cepas UPEC, no ha sido posible determinar un perfil de urovirulencia, dado que la mitad de todos los aislamientos de UPEC no contienen ninguno o solo uno de los determinantes de virulencia identificados. El objetivo de este estudio fue evaluar la relación entre determinantes de virulencia (serotipificación, capacidad de adherencia, producción de biofilm y toxinas) y perfil de resistencia con los grupos filogenéticos de E.coli en aislamientos de ITU de pacientes ambulatorios en 2 localidades en México.
MetodologíaAislamientos bacterianosDurante el periodo comprendido entre septiembre de 2010 y agosto de 2011 se recuperaron aislamientos clínicos de E.coli a partir de muestras de orina de pacientes con ITU adquiridas en la comunidad. Solo se trabajó una cepa por paciente, y fueron de cistitis aguda no complicada (pacientes ambulatorios, mujeres no embarazadas y sin ninguna otra enfermedad concomitante). Los urocultivos fueron procesados por métodos convencionales y se incluyeron las muestras con cuenta viable >105UFC/ml. Los aislamientos clínicos fueron identificados por el sistema semiautomatizado API20E (Biomérieux) y pruebas bioquímicas. Cincuenta cepas fueron recuperadas de la clínica número61 del Instituto Mexicano del Seguro Social (IMSS) y 57 del hospital del ISSSTE en Chilpancingo, Guerrero, ubicados al centro y al suroeste de México, respectivamente.
Pruebas de susceptibilidadLas pruebas de susceptibilidad a antibióticos se llevaron a cabo por el método de difusión en disco (Oxoid Ltd, Basingstoke, Reino Unido) siguiendo las indicaciones del Clinical and Laboratory Standars Institute17. Los aislamientos con resistencia a 3 o más clases de antibióticos fueron considerados multirresistentes (MDR).
Ensayo de hemolisinaLa capacidad hemolítica de los aislamientos fue evaluada por el método propuesto por Scheffer et al.18 en 1988, con algunas modificaciones. Se obtuvieron cultivos a una concentración de 6×108células. Se incubaron 0,1ml del cultivo con 0,9ml de una suspensión de eritrocitos de carnero al 2% en buffer (20mM CaCl2, 10mM Tris y 140mM NaCl, pH 7,4) durante 30min a 37°C; transcurrido el tiempo, los tubos fueron colocados en hielo y centrifugados. La liberación de hemoglobina fue determinada a 530nm y los resultados fueron expresados en porcentaje de lisis comparado con los eritrocitos lisados en agua destilada. Se establecieron terciles, clasificando la lisis en baja (>14,2%), moderada (14,13-23,3%) y alta (>23,4%).
Producción de biofilmLa capacidad de formación de biofilm fue determinada por el método cuantitativo en microplacas, descrito por George A. O’Toole en 201119. Las lecturas fueron realizadas a una densidad óptica de 570nm en un lector de Elisa (Awareness Stat Fax 2100). Para cuantificar el biofilm, se tomó en cuenta la densidad óptica del crecimiento y la del cristal violeta20.
SerotipificaciónLa determinación de los serogrupos se llevó a cabo en el Laboratorio Patógenos Entéricos del Departamento de Salud Pública de la Faculta de Medicina de la Universidad Nacional Autónoma de México, con antisueros prepararos en dicho laboratorio. Se utilizaron antisueros preparados en conejo contra los 187 antígenos somáticos y 56 sueros monovalentes de antígenos flagelares. Los aislamientos fueron cultivados en agar soya tripticasa para el antígeno somático y en medio semisólido de Craige para el antígeno flagelar21.
Ensayos de adherenciaEn cada uno de los 24 pozos de la placa se colocó una lenteja de vidrio y las células Vero se colocaron para formar la monocapa (a una concentración de 2,5×105), se colocó 1ml de volumen final conteniendo medio Mínimo Esencial de Eagle y una suspensión bacteriana de 2,5×108. Se incubó a 37°C durante 3h, se realizaron 3 lavados con PBS, se fijaron con metanol durante un minuto. Después se tiñeron con Giemsa durante 20min y se lavaron 3 veces con agua destilada. Se agregó una mezcla acetona/xileno (50/50) durante un minuto. Para observar al microscopio se montaron con resina las lentejas con las células teñidas en un portaobjetos y se observaron a inmersión. Se consideró adhesión positiva cuando se encontró por lo menos el 10% de las células con más de 10 bacterias adheridas.
Extracción de ADNSe utilizó el kit InstaGene Matrix™ (Bio-Rad Laboratories, Hercules, CA, EE.UU.), siguiendo las instrucciones del fabricante.
Determinación de grupos filogenéticosLos grupos filogenéticos se determinaron por medio de una PCR múltiple para definir los 7 grupos reconocidos (A, B1, B2, C, D, E, F) y el clado1, descrita previamente en 2013 por Clermont et al.13. Se usó una Taq polimerasa (Life Technologies) y la PCR fue realizada en un termociclador Gene Amp® PCR System 2400 (Applied Biosystems, EE.UU.).
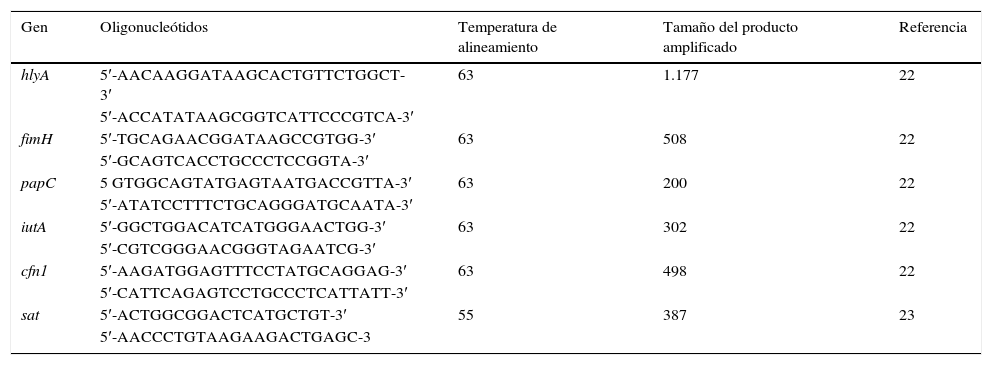
Detección de genes de virulenciaSe utilizó una PCR múltiple para los genes hlyA, fimH, papC, iutA, cnf1 y una PCR convencional simple para el gen sat. Los oligonucleótidos utilizados se muestran en la tabla 122,23. La mezcla de la reacción múltiple fue de 25μl, conteniendo 2μl de ADN, 4mM de MgCl2, 0,08mM de cada uno de los 4 dNTPs, 0,6μM de cada oligonucleótido y 2,5U de Taq polimerasa. Las condiciones de amplificación fueron: desnaturalización por 5min a 94°C, 25 ciclos de 30s a 94°C, 63°C/30s y 68°C/3min, y una extensión final a 72°C/10min. Para el gen sat la mezcla de reacción contuvo 3μl del buffer (10X), 0,9μl de MgCl2 (50mM), 0,6μl de dNTPs (2mM), 3μl (10μM) de cada oligonucleótido, 0,15μl de Taq ADN polimerasa (5U/μl), 3μl del templado de ADN y 16,35μl de H2O para dar un volumen final de 25μl. Bajo las siguientes condiciones: desnaturalización por 3min a 94°C, 35 ciclos de 1min a 94°C, alineamiento a 58°C 1min y una extensión final a 72°C/1min. Posteriormente se realizó la electroforesis en gel de agarosa (1,5%) a 100v durante 90min. Los geles fueron teñidos con bromuro de etidio y los amplificados fueron visualizados en un transiluminador ultravioleta.
Oligonucleótidos para la identificación de factores de virulencia
| Gen | Oligonucleótidos | Temperatura de alineamiento | Tamaño del producto amplificado | Referencia |
|---|---|---|---|---|
| hlyA | 5′-AACAAGGATAAGCACTGTTCTGGCT-3′ | 63 | 1.177 | 22 |
| 5′-ACCATATAAGCGGTCATTCCCGTCA-3′ | ||||
| fimH | 5′-TGCAGAACGGATAAGCCGTGG-3′ | 63 | 508 | 22 |
| 5′-GCAGTCACCTGCCCTCCGGTA-3′ | ||||
| papC | 5 GTGGCAGTATGAGTAATGACCGTTA-3′ | 63 | 200 | 22 |
| 5′-ATATCCTTTCTGCAGGGATGCAATA-3′ | ||||
| iutA | 5′-GGCTGGACATCATGGGAACTGG-3′ | 63 | 302 | 22 |
| 5′-CGTCGGGAACGGGTAGAATCG-3′ | ||||
| cfn1 | 5′-AAGATGGAGTTTCCTATGCAGGAG-3′ | 63 | 498 | 22 |
| 5′-CATTCAGAGTCCTGCCCTCATTATT-3′ | ||||
| sat | 5′-ACTGGCGGACTCATGCTGT-3′ | 55 | 387 | 23 |
| 5′-AACCCTGTAAGAAGACTGAGC-3 |
El ADN de los aislamientos con serogrupo O25 fue usados para realizar una PCR alelo específica para el gen pabB y determinar si las cepas pertenecen a la clona O25-ST13124.
Análisis estadísticoEl análisis estadístico se llevó a cabo utilizando el paquete estadístico STATA v.11.0. La distribución de las variables se reportó en base a sus frecuencias de aparición; las comparaciones de la producción de biofilm se realizaron con la prueba U de Mann-Whitney. Para comparar las frecuencias obtenidas de los genes de virulencia, adherencia y hemolisina se utilizó el test de chi cuadrado (χ2) y la prueba exacta de Fisher según su aplicabilidad. Se consideraron como significativos los valores de p≤0,05.
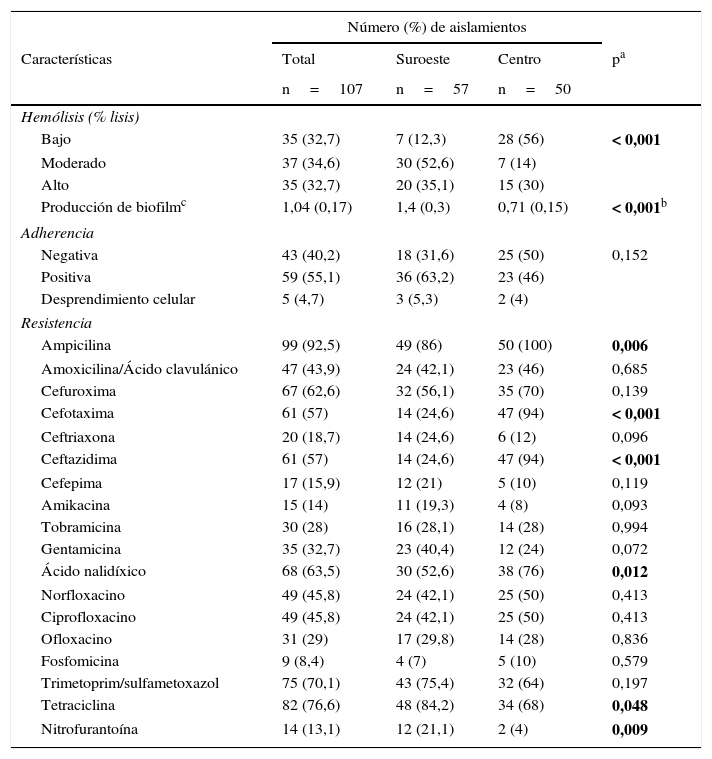
ResultadosCaracterísticas fenotípicasLas características fenotípicas evaluadas fueron: capacidad hemolítica, producción de biofilm, adherencia y resistencia antimicrobiana. Los aislamientos en la localidad al suroeste presentaron significativamente mayor capacidad hemolítica y producción de biofilm (p<0,001) en comparación con los aislamientos en la localidad en el centro del país. Los ensayos de adherencia en células Vero en cultivo mostraron que 59 cepas (55,1%) fueron positivas, 43 aislamientos (40,2%) resultaron negativos y 5 aislamientos (4,7%) ocasionaron desprendimiento celular. El patrón de adherencia en general fue de tipo agregativo.
El mayor porcentaje de resistencia se detectó frente a ampicilina (92,5%), tetraciclina (76,6%) y trimetoprim/sulfametoxazol (70,1%); se encontró diferencia estadísticamente significativa en la resistencia a ampicilina, cefotaxima, ceftazidima, ácido nalidíxico, tetraciclina y nitrofurantoína entre las 2 localidades, con mayor resistencia a ampicilina, cefotaxima, ceftazidima y ácido nalidíxico en los aislamientos provenientes de la localidad en el centro del país, en comparación con la localidad al suroeste, que presentó mayor resistencia a tetraciclina y nitrofurantoína (tabla 2).
Características fenotípicas de los aislamientos de Escherichia coli en 2 localidades en México
| Número (%) de aislamientos | ||||
|---|---|---|---|---|
| Características | Total | Suroeste | Centro | pa |
| n=107 | n=57 | n=50 | ||
| Hemólisis (% lisis) | ||||
| Bajo | 35 (32,7) | 7 (12,3) | 28 (56) | < 0,001 |
| Moderado | 37 (34,6) | 30 (52,6) | 7 (14) | |
| Alto | 35 (32,7) | 20 (35,1) | 15 (30) | |
| Producción de biofilmc | 1,04 (0,17) | 1,4 (0,3) | 0,71 (0,15) | < 0,001b |
| Adherencia | ||||
| Negativa | 43 (40,2) | 18 (31,6) | 25 (50) | 0,152 |
| Positiva | 59 (55,1) | 36 (63,2) | 23 (46) | |
| Desprendimiento celular | 5 (4,7) | 3 (5,3) | 2 (4) | |
| Resistencia | ||||
| Ampicilina | 99 (92,5) | 49 (86) | 50 (100) | 0,006 |
| Amoxicilina/Ácido clavulánico | 47 (43,9) | 24 (42,1) | 23 (46) | 0,685 |
| Cefuroxima | 67 (62,6) | 32 (56,1) | 35 (70) | 0,139 |
| Cefotaxima | 61 (57) | 14 (24,6) | 47 (94) | < 0,001 |
| Ceftriaxona | 20 (18,7) | 14 (24,6) | 6 (12) | 0,096 |
| Ceftazidima | 61 (57) | 14 (24,6) | 47 (94) | < 0,001 |
| Cefepima | 17 (15,9) | 12 (21) | 5 (10) | 0,119 |
| Amikacina | 15 (14) | 11 (19,3) | 4 (8) | 0,093 |
| Tobramicina | 30 (28) | 16 (28,1) | 14 (28) | 0,994 |
| Gentamicina | 35 (32,7) | 23 (40,4) | 12 (24) | 0,072 |
| Ácido nalidíxico | 68 (63,5) | 30 (52,6) | 38 (76) | 0,012 |
| Norfloxacino | 49 (45,8) | 24 (42,1) | 25 (50) | 0,413 |
| Ciprofloxacino | 49 (45,8) | 24 (42,1) | 25 (50) | 0,413 |
| Ofloxacino | 31 (29) | 17 (29,8) | 14 (28) | 0,836 |
| Fosfomicina | 9 (8,4) | 4 (7) | 5 (10) | 0,579 |
| Trimetoprim/sulfametoxazol | 75 (70,1) | 43 (75,4) | 32 (64) | 0,197 |
| Tetraciclina | 82 (76,6) | 48 (84,2) | 34 (68) | 0,048 |
| Nitrofurantoína | 14 (13,1) | 12 (21,1) | 2 (4) | 0,009 |
En negrita, los valores estadísticamente significativos.
Los resultados serológicos mostraron para el antígeno «O» que de 107 aislamientos analizados, 98 (91,5%) fueron tipificables, 9 (8,4%) fueron no tipificables y 9 aislamientos presentaron un fenotipo rugoso. Los aislamientos tipificables se distribuyeron en 43 serotipos diferentes, entre los cuales el serotipo más frecuente fue el O25:H4, con 23 (21,5%) aislamientos: 9 (15,7%) en la localidad al suroeste y 14 (28%) en la localidad en el centro del país. Para el antígeno «H» se encontró que 70 (65,4%) aislamientos fueron tipificables, 10 (9,3%) fueron no móviles y 27 aislamientos (25,2%) fueron no tipificables. Se encontró una diferencia estadísticamente significativa entre serotipo y localidad (p=0,002).
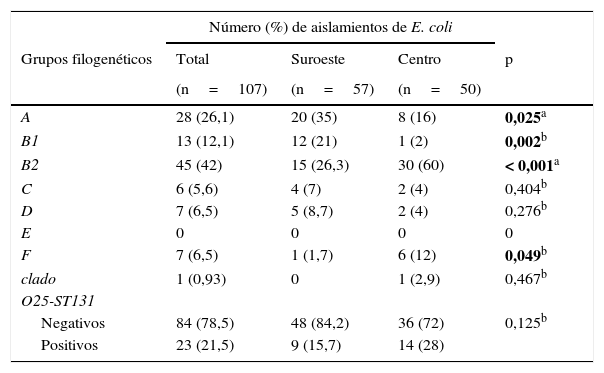
Grupos filogenéticosDel total de aislamientos el grupo B2 (42%) fue el más predominante, seguido del grupo A (26,1%). Los grupos A y B1 fueron más prevalentes (35 y 21%, respectivamente) en la localidad al suroeste, en contraste con los grupos filogenéticos B2 y F (60 y 12%, respectivamente), que fueron más frecuentes en la localidad en el centro del país, observándose diferencias estadísticamente significativas en estos grupos filogenéticos. El grupo filogenético E no fue detectado. También se detectó el grupo O25-ST131 con una frecuencia del 21,5%, siendo en su mayoría aislado en la comunidad del centro del país (tabla 3).
Distribución de los grupos filogenéticos y O25-ST131 en 2 localidades de México
| Número (%) de aislamientos de E. coli | ||||
|---|---|---|---|---|
| Grupos filogenéticos | Total | Suroeste | Centro | p |
| (n=107) | (n=57) | (n=50) | ||
| A | 28 (26,1) | 20 (35) | 8 (16) | 0,025a |
| B1 | 13 (12,1) | 12 (21) | 1 (2) | 0,002b |
| B2 | 45 (42) | 15 (26,3) | 30 (60) | < 0,001a |
| C | 6 (5,6) | 4 (7) | 2 (4) | 0,404b |
| D | 7 (6,5) | 5 (8,7) | 2 (4) | 0,276b |
| E | 0 | 0 | 0 | 0 |
| F | 7 (6,5) | 1 (1,7) | 6 (12) | 0,049b |
| clado | 1 (0,93) | 0 | 1 (2,9) | 0,467b |
| O25-ST131 | ||||
| Negativos | 84 (78,5) | 48 (84,2) | 36 (72) | 0,125b |
| Positivos | 23 (21,5) | 9 (15,7) | 14 (28) | |
En negrita, los valores estadísticamente significativos.
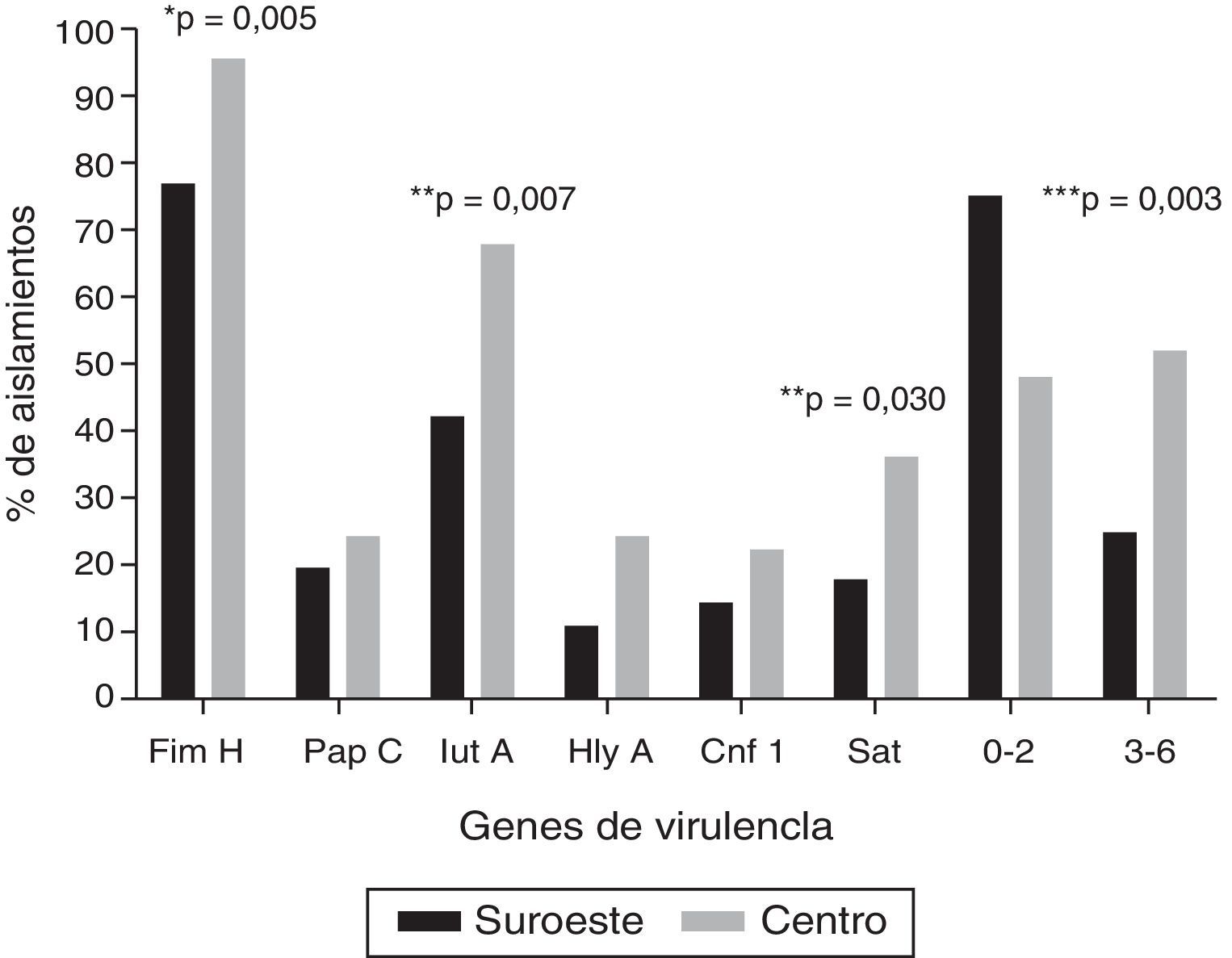
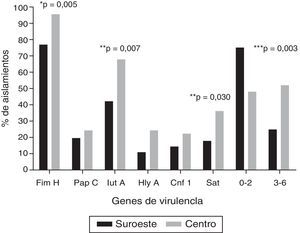
Los genes de virulencia más frecuentes, independientemente de la localización geográfica, fueron fimH (86%), seguido por iutA (54,2%) y sat (26,2%); 4 aislamientos (3,7%) fueron positivos a los 6 genes evaluados. La figura 1 muestra la distribución de los genes de virulencia entre las cepas UPEC analizadas en las 2 localidades: los 6 genes evaluados fueron encontrados en ambas localidades. La carga de genes fue mayor en los aislamientos en la localidad al centro en comparación con los aislamientos en la localidad del sureste (U-test, p=0,0152). Cabe resaltar que en 8 aislamientos (7,5%) no se encontró ninguno de los 6 genes de virulencia evaluados. En el ensayo de hemólisis todos los aislamientos positivos para hlyA presentaron actividad hemolítica.
Frecuencia de genes de virulencia en cepas de Escherichia coli uropatógena en 2 localidades en México.
* Prueba exacta de Fisher.
** Prueba de χ2. Únicamente se muestran los valores de p estadísticamente significativos (p≤0,05).
*** Relación entre los grupos que contienen de 0-2 genes y de 3-6 genes de virulencia.
No se encontró asociación entre la presencia de genes de virulencia y la producción de biopelícula (datos no mostrados). Sin embargo, papC y sat se asociaron de manera significativa con la adherencia a células Vero en cultivo (p<0,001 y p=0,010, respectivamente).
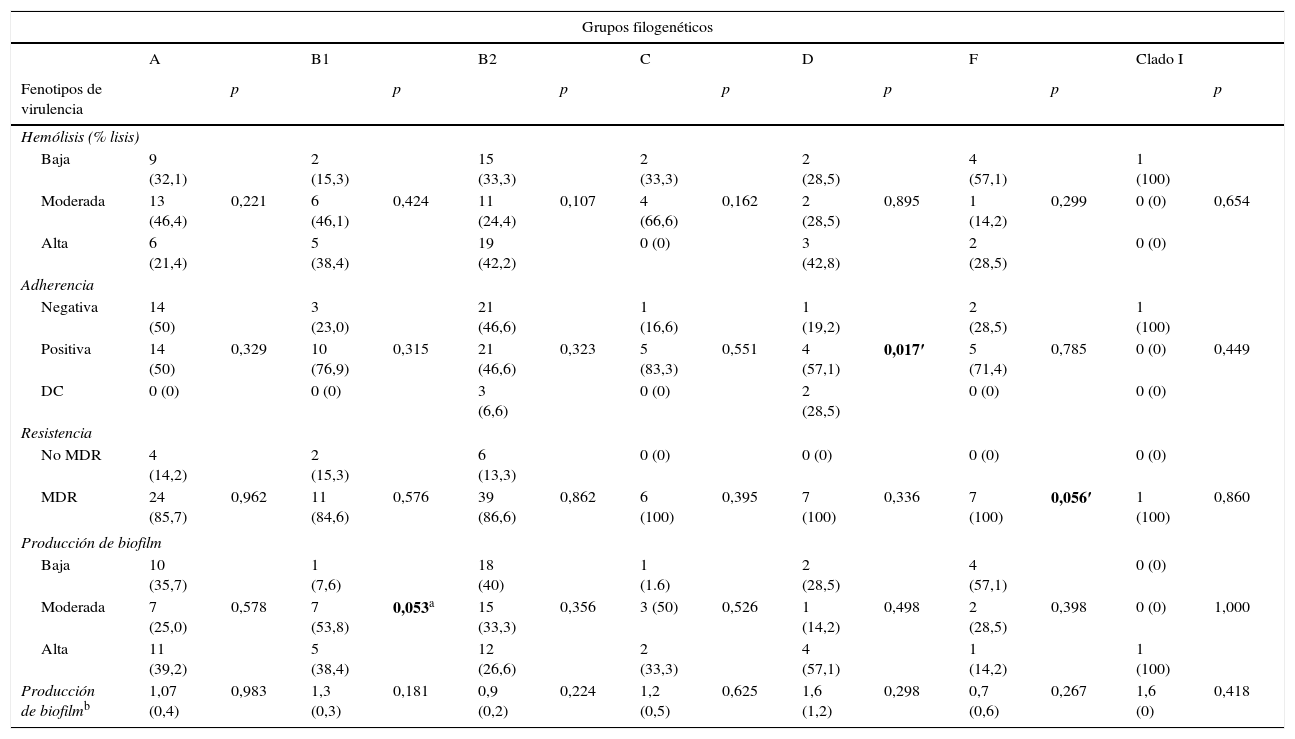
Factores de virulencia, grupo filogenético y resistencia antimicrobianaAl correlacionar los determinantes fenotípicos de virulencia con los grupos filogenéticos se encontró mayor producción de hemólisis en los grupos B2 y D (42,2 y 42,8%, respectivamente). Los ensayos de adherencia mostraron que todos los grupos filogenéticos fueron capaces de adherirse a células Vero, a excepción del cladoi. Los porcentajes de adherencia fueron mayores al 50% en cada grupo, y en el grupo D esta asociación fue estadísticamente significativa (p=0,017).
Del total de aislamientos, 92 (86%) fueron clasificados como MDR; dichos aislamientos pertenecieron a todos los grupos filogenéticos determinados, con frecuencias mayores al 85%. La MDR estuvo asociada significativamente con el grupo filogenético F (p=0,056). La producción de biofilm fue clasificada en baja, moderada y alta dependiendo de la densidad óptica. La producción de biofilm alta fue en los grupos filogenéticos A y B1, y la producción de biofilm fue asociada significativamente con el grupo B1 (p=0,053). Cabe mencionar que el 86% de los aislamientos productores de biofilm fueron multirresistentes y el serotipo O25:H4 fue el más MDR (tabla 4).
Relación entre grupo filogenético con el fenotipo de virulencia y multirresistencia en Escherichia coli uropatógena
| Grupos filogenéticos | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | B1 | B2 | C | D | F | Clado I | ||||||||
| Fenotipos de virulencia | p | p | p | p | p | p | p | |||||||
| Hemólisis (% lisis) | ||||||||||||||
| Baja | 9 (32,1) | 2 (15,3) | 15 (33,3) | 2 (33,3) | 2 (28,5) | 4 (57,1) | 1 (100) | |||||||
| Moderada | 13 (46,4) | 0,221 | 6 (46,1) | 0,424 | 11 (24,4) | 0,107 | 4 (66,6) | 0,162 | 2 (28,5) | 0,895 | 1 (14,2) | 0,299 | 0 (0) | 0,654 |
| Alta | 6 (21,4) | 5 (38,4) | 19 (42,2) | 0 (0) | 3 (42,8) | 2 (28,5) | 0 (0) | |||||||
| Adherencia | ||||||||||||||
| Negativa | 14 (50) | 3 (23,0) | 21 (46,6) | 1 (16,6) | 1 (19,2) | 2 (28,5) | 1 (100) | |||||||
| Positiva | 14 (50) | 0,329 | 10 (76,9) | 0,315 | 21 (46,6) | 0,323 | 5 (83,3) | 0,551 | 4 (57,1) | 0,017′ | 5 (71,4) | 0,785 | 0 (0) | 0,449 |
| DC | 0 (0) | 0 (0) | 3 (6,6) | 0 (0) | 2 (28,5) | 0 (0) | 0 (0) | |||||||
| Resistencia | ||||||||||||||
| No MDR | 4 (14,2) | 2 (15,3) | 6 (13,3) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | |||||||
| MDR | 24 (85,7) | 0,962 | 11 (84,6) | 0,576 | 39 (86,6) | 0,862 | 6 (100) | 0,395 | 7 (100) | 0,336 | 7 (100) | 0,056′ | 1 (100) | 0,860 |
| Producción de biofilm | ||||||||||||||
| Baja | 10 (35,7) | 1 (7,6) | 18 (40) | 1 (1.6) | 2 (28,5) | 4 (57,1) | 0 (0) | |||||||
| Moderada | 7 (25,0) | 0,578 | 7 (53,8) | 0,053a | 15 (33,3) | 0,356 | 3 (50) | 0,526 | 1 (14,2) | 0,498 | 2 (28,5) | 0,398 | 0 (0) | 1,000 |
| Alta | 11 (39,2) | 5 (38,4) | 12 (26,6) | 2 (33,3) | 4 (57,1) | 1 (14,2) | 1 (100) | |||||||
| Producción de biofilmb | 1,07 (0,4) | 0,983 | 1,3 (0,3) | 0,181 | 0,9 (0,2) | 0,224 | 1,2 (0,5) | 0,625 | 1,6 (1,2) | 0,298 | 0,7 (0,6) | 0,267 | 1,6 (0) | 0,418 |
DC: desprendimiento celular.
En negrita, los valores estadísticamente significativos.
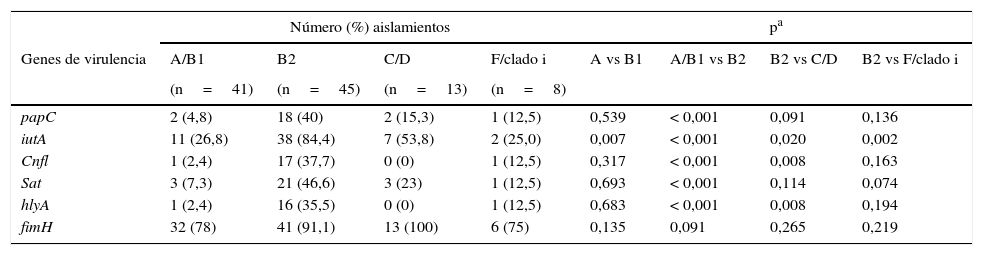
La mayor cantidad de genes de virulencia evaluados pertenecieron al grupo filogenético B2 (tabla 5). El gen fimH se distribuyó en todos los grupos. Se encontró mayor frecuencia de los genes papC y sat en el grupo B2 en comparación con los grupos A/B1. El gen iutA fue significativamente más frecuente en el grupo B2 en comparación los otros grupos filogenéticos. Los genes cnfl y hlyA fueron más frecuentes en el grupo B2 en comparación el grupo A, B1, C y D.
Distribución de genes de virulencia entre los grupos filogenéticos de Escherichia coli uropatógena
| Número (%) aislamientos | pa | |||||||
|---|---|---|---|---|---|---|---|---|
| Genes de virulencia | A/B1 | B2 | C/D | F/clado i | A vs B1 | A/B1 vs B2 | B2 vs C/D | B2 vs F/clado i |
| (n=41) | (n=45) | (n=13) | (n=8) | |||||
| papC | 2 (4,8) | 18 (40) | 2 (15,3) | 1 (12,5) | 0,539 | < 0,001 | 0,091 | 0,136 |
| iutA | 11 (26,8) | 38 (84,4) | 7 (53,8) | 2 (25,0) | 0,007 | < 0,001 | 0,020 | 0,002 |
| Cnfl | 1 (2,4) | 17 (37,7) | 0 (0) | 1 (12,5) | 0,317 | < 0,001 | 0,008 | 0,163 |
| Sat | 3 (7,3) | 21 (46,6) | 3 (23) | 1 (12,5) | 0,693 | < 0,001 | 0,114 | 0,074 |
| hlyA | 1 (2,4) | 16 (35,5) | 0 (0) | 1 (12,5) | 0,683 | < 0,001 | 0,008 | 0,194 |
| fimH | 32 (78) | 41 (91,1) | 13 (100) | 6 (75) | 0,135 | 0,091 | 0,265 | 0,219 |
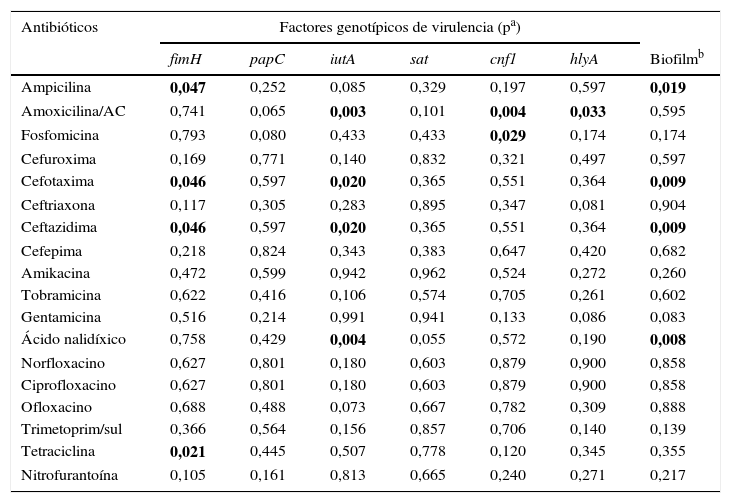
La tabla 6 muestra la relación entre resistencia antimicrobiana y genes de virulencia. La frecuencia del gen fimH fue significativamente mayor en los aislamientos resistentes a ampicilina, cefotaxima, ceftazidima y tetraciclina en comparación con los aislamientos susceptibles. El gen iutA se asoció con la resistencia a amoxicilina/ácido clavulánico, cefotaxima, ceftazidima y ácido nalidíxico. El gen cnf1 se asoció con la resistencia amoxicilina/ácido clavulánico y a fosfomicina (valores de p=0,004 y p=0,029, respectivamente). El gen hlyA solo se asoció con amoxicilina/ácido clavulánico (p=0,033). Cuando se analizó la producción de biofilm con la resistencia antimicrobiana se encontró una diferencia estadísticamente significativa con la resistencia a ampicilina, cefotaxima, ceftazidima y ácido nalidíxico. Al evaluar la resistencia y el grupo filogenético se encontró que los aislamientos pertenecientes al grupo filogenético B2 fueron más resistentes a cefotaxima y ceftazidima (p=0,009).
Relación entre factores de virulencia y resistencia a antimicrobianos
| Antibióticos | Factores genotípicos de virulencia (pa) | ||||||
|---|---|---|---|---|---|---|---|
| fimH | papC | iutA | sat | cnf1 | hlyA | Biofilmb | |
| Ampicilina | 0,047 | 0,252 | 0,085 | 0,329 | 0,197 | 0,597 | 0,019 |
| Amoxicilina/AC | 0,741 | 0,065 | 0,003 | 0,101 | 0,004 | 0,033 | 0,595 |
| Fosfomicina | 0,793 | 0,080 | 0,433 | 0,433 | 0,029 | 0,174 | 0,174 |
| Cefuroxima | 0,169 | 0,771 | 0,140 | 0,832 | 0,321 | 0,497 | 0,597 |
| Cefotaxima | 0,046 | 0,597 | 0,020 | 0,365 | 0,551 | 0,364 | 0,009 |
| Ceftriaxona | 0,117 | 0,305 | 0,283 | 0,895 | 0,347 | 0,081 | 0,904 |
| Ceftazidima | 0,046 | 0,597 | 0,020 | 0,365 | 0,551 | 0,364 | 0,009 |
| Cefepima | 0,218 | 0,824 | 0,343 | 0,383 | 0,647 | 0,420 | 0,682 |
| Amikacina | 0,472 | 0,599 | 0,942 | 0,962 | 0,524 | 0,272 | 0,260 |
| Tobramicina | 0,622 | 0,416 | 0,106 | 0,574 | 0,705 | 0,261 | 0,602 |
| Gentamicina | 0,516 | 0,214 | 0,991 | 0,941 | 0,133 | 0,086 | 0,083 |
| Ácido nalidíxico | 0,758 | 0,429 | 0,004 | 0,055 | 0,572 | 0,190 | 0,008 |
| Norfloxacino | 0,627 | 0,801 | 0,180 | 0,603 | 0,879 | 0,900 | 0,858 |
| Ciprofloxacino | 0,627 | 0,801 | 0,180 | 0,603 | 0,879 | 0,900 | 0,858 |
| Ofloxacino | 0,688 | 0,488 | 0,073 | 0,667 | 0,782 | 0,309 | 0,888 |
| Trimetoprim/sul | 0,366 | 0,564 | 0,156 | 0,857 | 0,706 | 0,140 | 0,139 |
| Tetraciclina | 0,021 | 0,445 | 0,507 | 0,778 | 0,120 | 0,345 | 0,355 |
| Nitrofurantoína | 0,105 | 0,161 | 0,813 | 0,665 | 0,240 | 0,271 | 0,217 |
AC: ácido clavulánico; SxT: trimetoprim-sulfametoxazol.
En negrita, los valores estadísticamente significativos.
Se ha reportado que las cepas de E.coli causantes de ITU pertenecen en su gran mayoría a los grupos filogenéticos B2 y D y poseen mayor cantidad de determinantes de virulencia en comparación con las cepas consideradas comensales pertenecientes a los grupos filogenéticos A y B122. En nuestro estudio, en la localidad del centro del país la mayoría de los aislamientos pertenecieron al grupo filogenético B2 (60%) y presentaron mayor cantidad de determinantes de virulencia; en contraste, en la localidad al suroeste del país el grupo filogenético A fue el más frecuente (35%) y presentó menor cantidad de genes de virulencia. También el grupo F fue más prevalente en la localidad del centro (valor estadísticamente significativo), mientras que el C y el D fueron más prevalentes en el suroeste, aunque no se encontraron diferencias significativas. En un estudio previo realizado en México25, donde solo se determinaron 4 grupos filogenéticos, se encontró que la mayoría de los aislamientos asociados con ITU pertenecen al grupo filogenético B2 (55,6%), seguido del grupo A (30,6%). Resultados similares fueron reportados en la región de Faisalabad, en Pakistán26: el 50% de los aislamientos de UPEC pertenecieron al grupo B2, seguido de los grupos A y B1 (19%). Nuestros resultados confirman que cepas consideradas comensales (grupos A y B1) pueden ocasionar ITU, y estas cepas pueden adquirir genes de virulencia en el tracto gastrointestinal por transferencia horizontal que les permita colonizar el tracto urinario12,25. Además, estudios realizados en cepas comensales y patógenas en diferentes regiones indican que las condiciones geográficas/climáticas, la dieta, el uso de antibióticos y los factores genéticos pueden influir en la flora comensal para adquirir genes de virulencia y volverse potencialmente patógenas27.
Se han documentado los mecanismos por medio de los cuales los factores de virulencia promueven el desarrollo de ITU, y el número de genes puede ser proporcional al potencial patógeno12,28; los genes fimH, iutA y sat (96, 68 y 36%, respectivamente) fueron más frecuentes en la localidad en el centro, donde el grupo filogenético B fue más predominante. La alta frecuencia del gen de fimH indica su importancia en la etapa inicial de la infección, ya que FimH reconoce a los receptores sobre la superficie de las células de la vejiga, facilitando la colonización bacteriana, mientras que IutA es el receptor hidroxamato más importante durante la infección que contribuye a la colonización29,30.
La toxina Sat es una serina-proteasa que exhibe actividad citopática sobre células HEp-2 y Vero; a nivel renal se ha caracterizado por provocar vacuolización y daño glomerular28. PapC es una proteína de membrana externa esencial para la biogénesis del pili P, el cual se ha asociado a pielonefritis por la adherencia de la adhesina PapG a los receptores sobre la célula huésped8. En este estudio los genes sat y papC se asociaron de manera significativa con la adherencia a células Vero, lo cual puede ser indicativo de que estas cepas causen infecciones en vías urinarias altas. El patrón de adherencia observado de manera general fue de tipo agregativo, el cual es característico de E.coli enteroagregativa (EAEC). Abe et al.31 reportaron este fenotipo en el 6,9% de las cepas UPEC adherentes y mencionan que es posible que algunos aislamientos de EAEC causen ITU, por lo que sería importante determinar características de virulencia de patotipos diarreogénicos en cepas UPEC.
Algunos estudios reportan a la α-hemolisina como la toxina más prevalente15,32. La frecuencia de hlyA y cnf1 (16,8 y 17,8%, respectivamente) fue mayor en nuestro estudio en comparación a reportes previos en México realizados por López-Banda et al.25, en donde la prevalencia registrada fue del 7,4 y del 6,5%, respectivamente. La frecuencia de aislamientos con un porcentaje de hemólisis alto pertenecieron al grupo filogenético B2, lo que sugiere que la presencia de HlyA podría estar asociada con la severidad de las ITU28. La presencia combinada de papC, cnf1 y hlyA es evidencia de la isla de patogenicidad IIJ96, la cual ya ha sido reportada en cepas de E.coli causantes de ITU33. En nuestro estudio, la presencia simultánea de papC, cnf1 y hlyA fue observada en 9 aislamientos; esta frecuencia es mayor a la reportada en otros países, que va de 2 a 4 aislamientos25,26,32.
La resistencia a antibióticos en este estudio fue mayor a lo reportado en México y en otros países25,32,34,35. Las diferencias significativas en la resistencia a ampicilina, cefotaxima, ceftazidima y ácido nalidíxico entre las poblaciones estudiadas pueden ser debidas a que en la localidad del centro del país la mayoría de los aislamientos pertenecieron al grupo filogenético B2. El grupo filogenético B se ha relacionado con el grupo O25bST131 (el 21,5% de los aislamientos MDR pertenecieron a este grupo), el cual ya ha logrado extenderse en México16. El grupo O25bST131 se ha asociado con la producción de β-lactamasas de espectro extendido (principalmente tipo CTX-M-15) y resistencia a fluoroquinolonas en la comunidad24,36. Además, se encontró una asociación estadísticamente significativa de estos 3 antibióticos con la producción de biofilm, lo que sugiere que la adquisición de resistencia a algunos antibióticos puede disminuir la producción de biofilm, como en el caso de la resistencia a cefalosporinas de tercera generación y quinolonas37,38. Se ha documentado también que las bacterias formadoras de biofilm presentan un incremento en la resistencia a antimicrobianos y a la cronicidad de la infección, además de estar asociado con ITU recurrentes39,40. Los aislamientos en la localidad del suroeste presentaron mayor resistencia a tetraciclina y nitrofurantoína (diferencias estadísticamente significativas) que la del centro del país. En México se ha reportado resistencia a nitrofurantoína del 5,1 y del 7,4%16,34. Sin embargo, nosotros encontramos que el 13,1% de los aislamientos fueron resistentes, y este incremento puede estar asociado al uso más frecuente de este antibiótico en la comunidad del sureste.
La resistencia a cefotaxima y ceftazidima puede estar relacionada con la producción de β-lactamasas de espectro extendido, las cuales pueden estar presentes el grupo filogenético A y B1 de la localidad del sureste.
En nuestro estudio la resistencia a quinolonas estuvo asociada con la presencia de genes iutA y sat; estos determinantes de virulencia han sido reportados en aislamientos resistentes a ácido nalidíxico32,41. La resistencia a amoxicilina/ácido clavulánico estuvo asociada con los genes iutA, cnf1 y hlyA; estos genes de virulencia han sido reportados en cepas UPEC productoras de β-lactamasas de espectro extendido36. Estos resultados reflejan la heterogeneidad en la distribución de genes de virulencia y resistencia a antibióticos entre las cepas UPEC; es importante describir las características que definen a los aislamientos de UPEC dentro de las diferentes poblaciones de estudio.
En conclusión, el grupo filogenético, los factores de virulencia y la susceptibilidad a antibióticos de E.coli causante de infecciones del ITU varían significativamente entre las poblaciones mexicanas estudiadas. El grupo filogenético A puede ser multirresistente y tiene la capacidad de producir infecciones urinarias. La MDR está presente en todos grupos filogenéticos, lo que complica y disminuye las opciones terapéuticas en pacientes con ITU en la comunidad.
FinanciaciónEste trabajo contó con el apoyo del proyecto financiado en la Convocatoria de Apoyo al Fortalecimiento y Desarrollo de la Infraestructura Científica y Tecnológica 2014, clave 229958. Asimismo se agradece el apoyo económico por parte del CONACYT al proyecto número 166004: «Papel de plásmidos e integrones en la multirresistencia a antimicrobianos en cepas de Escherichia coli uropatógena y su posible asociación con la adherencia e invasividad en células cultivadas».
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Al Laboratorio de Patógenos Entéricos del departamento de Salud Pública de la Facultad de Medicina de la UNAM, por la serotipificación de las cepas.