En los últimos años se ha observado un incremento de la resistencia a fluoroquinolonas en enterobacterias, estando asociado significativamente a la resistencia a betalactámicos. Nuestro objetivo fue conocer la prevalencia de mecanismos cromosómicos y plasmídicos de resistencia a quinolonas en aislados productores de betalactamasas de claseC adquiridas y/o carbapenemasas.
MétodosSe evaluó la presencia de mecanismos cromosómicos y plasmídicos de resistencia a quinolonas [mutaciones en la región determinante de resistencia a quinolonas de gyrA y parC y genes qnr, aac(6′)-Ib-cr y qepA] en 289 aislados de enterobacterias productoras de betalactamasas de claseC adquiridas y/o carbapenemasas recogidos entre febrero y julio de 2009 en 35 hospitales españoles.
ResultadosSe detectaron determinantes plasmídicos en 92 aislados (31,8%); en 83 aislados (28,7%) se detectó algún gen qnr, y en 20 (7%), la variante aac(6′)-Ib-cr. El gen qnr más prevalente fue qnrB4 (20%), asociado en la mayoría de los casos a DHA-1. El 14,6% de los aislados con una CMI de ciprofloxacino superior a 0,25mg/l no presentaban mutaciones en gyrA ni parC, detectándose en el 90% de los mismos algún determinante plasmídico de resistencia a quinolonas.
ConclusiónqnrB4 fue el determinante plasmídico más prevalente, claramente asociado a DHA-1. Los mecanismos plasmídicos en asociación con mecanismos cromosómicos diferentes a las mutaciones en los genes de las topoisomerasas (sobreexpresión de bombas de expulsión, alteración del lipopolisacárido o disminución de porinas) pueden dar lugar a valores de CMI de ciprofloxacino que superan los puntos de corte establecidos por los principales comités internacionales de definición de puntos de corte para interpretación de datos de sensibilidad.
Quinolone resistance in Enterobacteriaceae species has increased over the past few years, and is significantly associated to beta-lactam resistance. The aim of this study was to evaluate the prevalence of chromosomal- and plasmid-mediated quinolone resistance in acquired AmpC β-lactamase and/or carbapenemase-producing Enterobacteriaceae isolates.
MethodsThe presence of chromosomal- and plasmid-mediated quinolone resistance mechanisms [mutations in the quinolone resistance determining region (QRDR) of gyrA and parC and qnr, aac(6′)-Ib-cr and qepA genes] was evaluated in 289 isolates of acquired AmpC β-lactamase- and/or carbapenemase-producing Enterobacteriaceae collected between February and July 2009 in 35 Spanish hospitals.
ResultsPlasmid mediated quinolone resistance (PMQR) genes were detected in 92 isolates (31.8%), qnr genes were detected in 83 isolates (28.7%), and the aac(6′)-Ib-cr gene was detected in 20 isolates (7%). qnrB4 gene was the most prevalent qnr gene detected (20%), associated, in most cases, with DHA-1. Only 14.6% of isolates showed no mutations in gyrA or parC with a ciprofloxacin MIC of 0.5mg/L or higher, whereas PMQR genes were detected in 90% of such isolates.
ConclusionqnrB4 gene was the most prevalent PMQR gene detected, and was significantly associated with acquired AmpC β-lactamase DHA-1. PMQR determinants in association with other chromosomal-mediated quinolone resistance mechanisms, different to mutations in gyrA and parC (increased energy-dependent efflux, altered lipopolysaccharide or porin loss), could lead to ciprofloxacin MIC values that exceed breakpoints established by the main international committees to define clinical antimicrobial susceptibility breakpoints.
Las fluoroquinolonas son antimicrobianos de amplio espectro que se utilizan con elevada frecuencia en clínica. Desde hace años se están observando altas tasas de resistencia a estos antimicrobianos en enterobacterias en todo el mundo1. Las dianas de estos compuestos son la topoisomerasaii (o ADN girasa) y la topoisomerasaiv. La resistencia a quinolonas en enterobacterias puede deberse a múltiples causas que con frecuencia se expresan en un mismo microorganismo. Tradicionalmente se ha prestado especial atención a las mutaciones que ocurren en los genes gyrA y parC que codifican la subunidad A de la ADN girasa y la subunidad A de la topoisomerasaiv, respectivamente; menos relevantes en aislamientos clínicos son las mutaciones en los genes gyrB y parE, que codifican las subunidades B de las citadas enzimas. En estos genes, las mutaciones asociadas a resistencia a quinolonas se localizan en una región concreta de los mismos denominada «región determinante de la resistencia a quinolonas» (quinolone resistance determining region [QRDR]). En las bacterias gramnegativas la diana primaria de las quinolonas es la ADN girasa, apareciendo las mutaciones primero en gyrA y posteriormente en parC1. A pesar de ello, se han descrito aislados clínicos resistentes a ciprofloxacino con mutaciones en parC que no se acompañan de mutaciones en gyrA2. Una única mutación en gyrA causa resistencia a ácido nalidíxico, pero confiere un bajo nivel de resistencia (sin sobrepasar el punto de corte clínico) a ciprofloxacino y levofloxacino3. Al aumentar el número de mutaciones en gyrA y parC, lo hace también el nivel de resistencia hasta rebasar el valor que define la resistencia clínica. Aunque en menor medida, la pérdida o la alteración estructural de las porinas, la sobreexpresión de bombas de expulsión activa y las alteraciones del lipopolisacárido también contribuyen a la resistencia a quinolonas4.
Hasta la fecha se han descrito 3 tipos de determinantes plasmídicos de resistencia a quinolonas (DPRQ): los genes qnr, que codifican proteínas de repeticiones pentapeptídicas; la variante de una acetiltransferasa, AAC(6′)-Ib-cr, que afecta a quinolonas con un grupo piperazinil (por ejemplo, ciprofloxacino y norfloxacino), y las bombas de expulsión activa OqxAB (muy frecuente en el cromosoma de Klebsiella pneumoniae) y QepA5. En las 2 últimas décadas se está observado un aumento a nivel global de la prevalencia de los DPRQ4.
La resistencia a quinolonas es más frecuente en enterobacterias que presentan resistencia a betalactámicos por producción de betalactamasas de espectro extendido (BLEE), betalactamasas de claseC adquiridas o carbapenemasas6. De hecho, la primera descripción de un gen qnr (qnrA1) tuvo lugar en una cepa de K.pneumoniae productora de la cafalosporinasa FOX-57. Desde entonces, múltiples estudios han demostrado la frecuente asociación entre genes que codifican las citadas betalactamasas y diversas variantes de genes que codifican DPRQ, por ejemplo el caso de qnrA y qnrB con BLEE del grupo CTX-M4,8.
En el presente estudio se analiza la presencia de mutaciones cromosómicas (en gyrA y parC) y de los DPRQ qnrA, qnrB, qnrC, qnrD, qnrS, qepA y aac(6′)-Ib-cr en enterobacterias productoras de betalactamasas de claseC adquiridas y/o carbapenemasas procedentes de muestras clínicas, aisladas en un estudio multicéntrico en el que participaron 35 hospitales españoles que tuvo lugar entre febrero y julio de 20099. El objetivo es estudiar la asociación de mecanismos de resistencia a quinolonas (cromosómicos y plasmídicos) con betalactamasas de claseC adquiridas y/o carbapenemasas en enterobacterias, así como conocer la prevalencia de DPRQ en esta población bacteriana.
MétodosAislados bacterianosSe analizó una colección de 289 aislados de enterobacterias productoras de betalactamasas de claseC adquiridas y/o carbapenemasas, incluyendo Escherichia coli (n=163), K.pneumoniae (n=57), Proteus mirabilis (n=45), Klebsiella oxytoca (n=10), Enterobacter cloacae (n=9), Citrobacter koseri (n=3), Proteus penneri (n=1) y Salmonella sp. (n=1). Solo se incluyó un aislado por paciente. Los datos clínicos y epidemiológicos de esta colección han sido descritos previamente9,10.
Sensibilidad antimicrobiana y estudio de mecanismos de resistencia a quinolonasLa sensibilidad a ácido nalidíxico y ciprofloxacino se determinó mediante los métodos estandarizados de difusión con discos y microdilución en caldo, respectivamente11. En las cepas resistentes a ácido nalidíxico (diámetro del halo de inhibición ≤13mm)11 o con una CMI de ciprofloxacino ≥0,25mg/l se analizaron las mutaciones en la región QRDR de gyrA empleando la metodología descrita previamente12. En las cepas en que no se detectaron mutaciones en gyrA se estudió de forma adicional la presencia de mutaciones en parC12.
Se analizó la presencia de los genes qnrA, qnrB, qnrC, qnrD, qnrS, qepA y aac(6′)-Ib en todas las cepas mediante PCR utilizando cebadores descritos previamente13. Los productos de amplificación se secuenciaron posteriormente.
ResultadosSe detectaron DPRQ en 92 (31,8%) de los 289 aislados de enterobacterias analizados mediante PCR y secuenciación. Los genes de resistencia plasmídica a quinolonas detectados incluyeron 2 qnrA1, 59 qnrB (58 qnrB4, 1 qnrB19), 10 qnrD, 16 qnrS (8 qnrS1, 8 qnrS2) y 20 aac(6′)-Ib-cr. No se detectaron en ningún aislado los determinantes qnrC ni qepA. Setenta y siete aislados (83,7%) presentaron un solo DPRQ. Los 15 aislados restantes (16,3%) presentaron 2 DPRQ: 6 aac(6′)-Ib-cr+qnrS2, 5 aac(6′)-Ib-cr+qnrB4, 3 qnrB4+qnrS1 y 1 qnrB4+qnrS2.
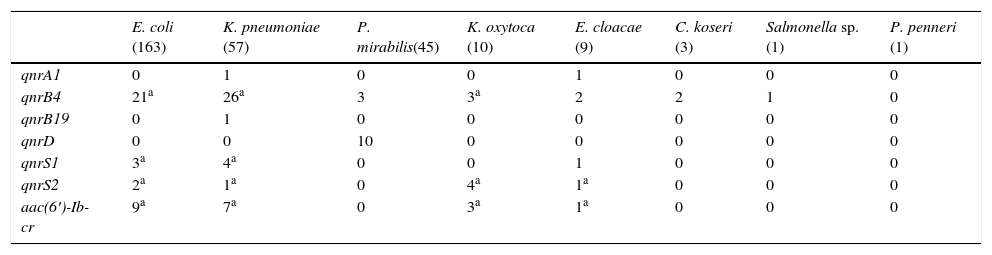
Se observaron importantes diferencias en el tipo de DPRQ acorde a las especies de enterobacterias (tabla 1). De los 163 aislados de E.coli, se detectó al menos un determinante plasmídico en 32 aislados (19,6%). Los determinantes detectados fueron: 21 qnrB4, 5 qnrS y 9 aac(6′)-Ib-cr, portando 3 aislados 2 DPRQ (2 qnrS2+aac(6′)-Ib-cr y 1 qnrB4+ qnrS1). Entre los aislados de K.pneumoniae se detectaron mecanismos plasmídicos en 32 (56,1%). En 24 se detectó un solo mecanismo plasmídico y en 8 se detectaron 2 (2 qnrB4+qnrS1, 5 qnrB4+aac(6′)-Ib-cr y 1 qnrS2+aac(6′)-Ib-cr). La prevalencia de DPRQ en el resto de especies fue: P.mirabilis 28,9%, K.oxytoca 70%, E.cloacae 55,6%, C.koseri 66,7%, Salmonella sp. 100% y P.penneri 0%. Los diferentes DPRQ detectados en estas especies se recogen en la tabla 1. El gen qnrD solo se detectó en P.mirabilis. El determinante qnrS2 fue detectado en presencia de otro gen plasmídico en 7 de los 8 aislados que lo portaban (87,5%).
Determinantes plasmídicos de resistencia a quinolonas detectados en función de la especie bacteriana
| E. coli (163) | K. pneumoniae (57) | P. mirabilis(45) | K. oxytoca (10) | E. cloacae (9) | C. koseri (3) | Salmonella sp. (1) | P. penneri (1) | |
|---|---|---|---|---|---|---|---|---|
| qnrA1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| qnrB4 | 21a | 26a | 3 | 3a | 2 | 2 | 1 | 0 |
| qnrB19 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| qnrD | 0 | 0 | 10 | 0 | 0 | 0 | 0 | 0 |
| qnrS1 | 3a | 4a | 0 | 0 | 1 | 0 | 0 | 0 |
| qnrS2 | 2a | 1a | 0 | 4a | 1a | 0 | 0 | 0 |
| aac(6′)-Ib-cr | 9a | 7a | 0 | 3a | 1a | 0 | 0 | 0 |
Entre los aislados productores de carbapenemasas (2 productores de IMP-22, un productor de IMP-28 y 25 productores de VIM-1) la prevalencia de DPRQ fue del 39%, detectándose algún DPRQ en 11 aislados productores de VIM-1: 2 qnrA1, 1 qnrB19, 1 qnrD, 1 qnrS1, 1 qnrS2, 1 qnrB4+aac(6′)-Ib-cr y 4 qnrS2+aac(6′)-Ib-cr.
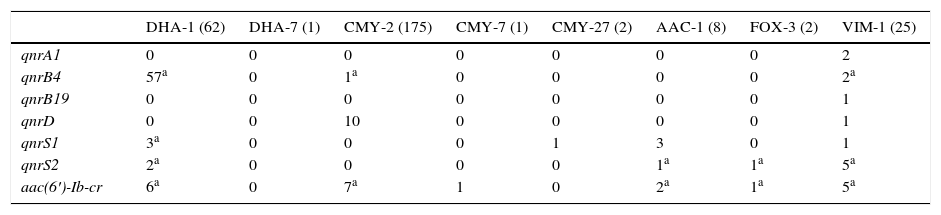
La prevalencia de mecanismos plasmídicos de resistencia a quinolonas entre los 264 aislados productores de betalactamasas de claseC adquiridas fue del 31,4% (83 aislados). No se detectaron determinantes plasmídicos en los aislados que producían las betalactamasas de caseC adquiridas DHA-6, FOX-8, CMY-4, CMY-48, CMY-55, CMY-57, CMY-59 y CMY-60. Los genes qnrA1 y qnrB19 no se detectaron en aislados productores de betalactamasas de claseC adquiridas. La asociación entre DPRQ y betalactamasas de claseC adquiridas se recoge en la tabla 2. La betalactamasa de claseC mayoritaria de la colección fue CMY-2 (175 aislados), detectándose en el 9,7% de los aislados portadores de esta enzima algún DPQR. Los 10 genes qnrD se detectaron en cepas de P.mirabilis productoras de CMY-2. Entre las enterobacterias productoras de CMY-2 se detectaron 7 con aac(6′)-Ib-cr.
Determinantes plasmídicos de resistencia a quinolonas detectados en función de la betalactamasa de clase C adquirida y carbapenemasa producida
| DHA-1 (62) | DHA-7 (1) | CMY-2 (175) | CMY-7 (1) | CMY-27 (2) | AAC-1 (8) | FOX-3 (2) | VIM-1 (25) | |
|---|---|---|---|---|---|---|---|---|
| qnrA1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| qnrB4 | 57a | 0 | 1a | 0 | 0 | 0 | 0 | 2a |
| qnrB19 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| qnrD | 0 | 0 | 10 | 0 | 0 | 0 | 0 | 1 |
| qnrS1 | 3a | 0 | 0 | 0 | 1 | 3 | 0 | 1 |
| qnrS2 | 2a | 0 | 0 | 0 | 0 | 1a | 1a | 5a |
| aac(6′)-Ib-cr | 6a | 0 | 7a | 1 | 0 | 2a | 1a | 5a |
No se incluyen aislados productores de las betalactamasas de case C adquiridas DHA-6, FOX-8, CMY-4, CMY-48, CMY-55, CMY-57, CMY-59 y CMY-60 y las carbapenemasas IMP-22 e IMP-28 porque en ninguno de ellos se detectaron DPRQ.
Entre los 62 aislados productores de DHA-1 se detectaron DPRQ en 58 (93,5%), siendo qnrB4 el más frecuentemente encontrado, presente en 57 de dichos aislados (en 9 aislados también se detectó un segundo determinante plasmídico). La asociación entre qnrB4 y DHA-1 fue estadísticamente significativa (p<0,05).
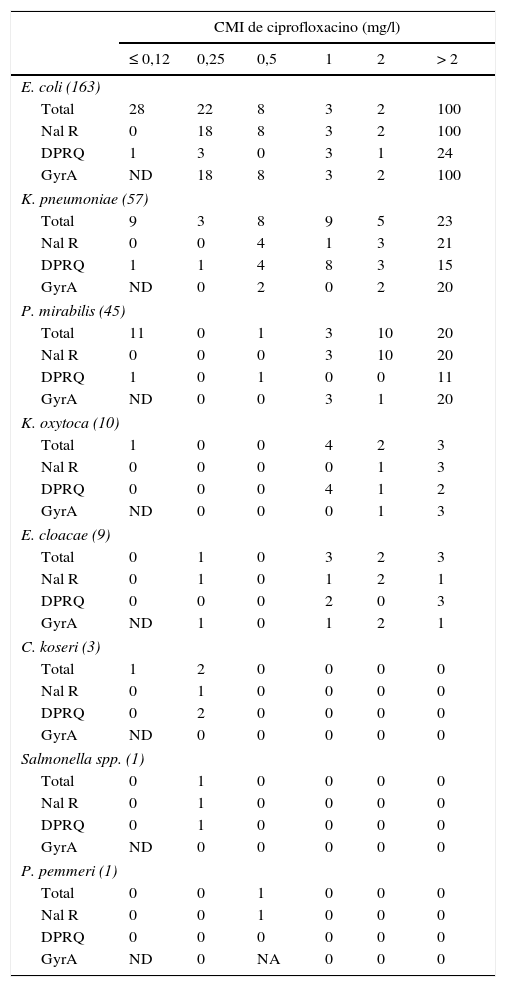
Con respecto a los mecanismos cromosómicos de resistencia a quinolonas, se analizó la región QRDR de gyrA de los aislados resistentes a ácido nalidíxico11 o que presentaban una CMI de ciprofloxacino igual o superior a 0,25mg/l. No se encontró ningún aislado con mutaciones en parC sin presentarla en gyrA. En E.coli se comprobó que todos los aislados resistentes a ácido nalidíxico presentaban mutaciones en la región analizada (tabla 3). En esta especie bacteriana hubo 4 aislados con CMI de ciprofloxacino de 0,25mg/l que no presentaron mutaciones en gyrA ni en parC; 3 de ellos presentaron un DPRQ. Además, en esta enterobacteria se encontraron aislados con mutaciones en gyrA y DPRQ independientemente del valor de su CMI de ciprofloxacino, algo no observado en los aislados de otras especies, en las cuales solo se detectó la combinación de DPRQ y mutaciones en gyrA en aislados con CMI de ciprofloxacino superior a 2mg/l.
Características de los aislados de la colección en función de la CMI de ciprofloxacino
| CMI de ciprofloxacino (mg/l) | ||||||
|---|---|---|---|---|---|---|
| ≤ 0,12 | 0,25 | 0,5 | 1 | 2 | > 2 | |
| E. coli (163) | ||||||
| Total | 28 | 22 | 8 | 3 | 2 | 100 |
| Nal R | 0 | 18 | 8 | 3 | 2 | 100 |
| DPRQ | 1 | 3 | 0 | 3 | 1 | 24 |
| GyrA | ND | 18 | 8 | 3 | 2 | 100 |
| K. pneumoniae (57) | ||||||
| Total | 9 | 3 | 8 | 9 | 5 | 23 |
| Nal R | 0 | 0 | 4 | 1 | 3 | 21 |
| DPRQ | 1 | 1 | 4 | 8 | 3 | 15 |
| GyrA | ND | 0 | 2 | 0 | 2 | 20 |
| P. mirabilis (45) | ||||||
| Total | 11 | 0 | 1 | 3 | 10 | 20 |
| Nal R | 0 | 0 | 0 | 3 | 10 | 20 |
| DPRQ | 1 | 0 | 1 | 0 | 0 | 11 |
| GyrA | ND | 0 | 0 | 3 | 1 | 20 |
| K. oxytoca (10) | ||||||
| Total | 1 | 0 | 0 | 4 | 2 | 3 |
| Nal R | 0 | 0 | 0 | 0 | 1 | 3 |
| DPRQ | 0 | 0 | 0 | 4 | 1 | 2 |
| GyrA | ND | 0 | 0 | 0 | 1 | 3 |
| E. cloacae (9) | ||||||
| Total | 0 | 1 | 0 | 3 | 2 | 3 |
| Nal R | 0 | 1 | 0 | 1 | 2 | 1 |
| DPRQ | 0 | 0 | 0 | 2 | 0 | 3 |
| GyrA | ND | 1 | 0 | 1 | 2 | 1 |
| C. koseri (3) | ||||||
| Total | 1 | 2 | 0 | 0 | 0 | 0 |
| Nal R | 0 | 1 | 0 | 0 | 0 | 0 |
| DPRQ | 0 | 2 | 0 | 0 | 0 | 0 |
| GyrA | ND | 0 | 0 | 0 | 0 | 0 |
| Salmonella spp. (1) | ||||||
| Total | 0 | 1 | 0 | 0 | 0 | 0 |
| Nal R | 0 | 1 | 0 | 0 | 0 | 0 |
| DPRQ | 0 | 1 | 0 | 0 | 0 | 0 |
| GyrA | ND | 0 | 0 | 0 | 0 | 0 |
| P. pemmeri (1) | ||||||
| Total | 0 | 0 | 1 | 0 | 0 | 0 |
| Nal R | 0 | 0 | 1 | 0 | 0 | 0 |
| DPRQ | 0 | 0 | 0 | 0 | 0 | 0 |
| GyrA | ND | 0 | NA | 0 | 0 | 0 |
DPRQ: número de aislados portadores de determinantes plasmídicos de resistencia a quinolonas; GyrA: número de aislados con mutaciones en la QRDR de gyrA. No se encontró ningún aislado sin mutaciones en gyrA que presentara alguna mutación en parC; NA: no amplificó la región QRDR de gyrA; Nal R: número de aislados resistentes a ácido nalidíxico (diámetro del halo ≤13mm); ND: no se determinó la presencia de mutaciones en gyrA en los aislados sensibles a ácido nalidíxico (diámetro del halo de inhibición >13mm)11 o con una CMI de ciprofloxacino ≤0,12mg/l; Total: número total de aislados de cada especie.
Entre los aislados de especies diferentes a E.coli, se encontró un 9,5% que eran resistentes a ácido nalidíxico y no presentaban mutaciones en el QRDR de gyrA ni de parC (tabla 3); el 66,7% de los aislados con una CMI de ciprofloxacino igual o superior a 0,25mg/l presentaban alguna mutación en gyrA y el 52,8% portaba algún DPRQ.
DiscusiónEl desarrollo y la expansión de la resistencia a los betalactámicos, unidos al amplio espectro antimicrobiano de las fluoroquinolonas, condujo al uso de estos últimos fármacos como terapia empírica para una gran variedad de infecciones de origen comunitario y nosocomial. El uso masivo de las quinolonas ha provocado un incremento de los niveles de resistencia a fluoroquinolonas a nivel mundial. En España, la tasa de aislados de E.coli resistentes a fluoroquinolonas ha aumentado en los últimos años más del 16%, pasando del 17,3% en 2001 al 34% en 201414. La situación en K.pneumoniae es similar, siendo el aumento de los niveles de resistencia algo inferior. En el presente estudio, el 59% (170) de los aislados portadores de carbapenemasas o betalactamasas de clase C adquiridas analizados fueron no sensibles a ciprofloxacino, dato preocupante que pone de manifiesto el reducido número de opciones terapéuticas que presentan los aislados de enterobacterias multirresistentes.
La resistencia a fluoroquinolonas se debe principalmente a mutaciones en los genes de las topoisomerasas tipoii y se trata de un proceso secuencial, de modo que la aparición de una primera mutación en gyrA favorece la aparición de nuevas mutaciones en parC y gyrA que conllevan a un incremento de la CMI de ciprofloxacino por encima de 2mg/l15. El resto de mecanismos de resistencia conocidos, tanto mecanismos cromosómicos como plasmídicos, confieren por sí solos un bajo nivel de resistencia que no llega a superar los puntos de corte establecidos por las agencias internacionales5. En la colección analizada en este estudio se han encontrado 24 aislados (15K.pneumoniae, 5K.oxytoca y 4E.cloacae) con una CMI de ciprofloxacino igual o superior a 1mg/l que no presentan mutaciones en la región QRDR de gyrA ni parC; 23 de estos aislados presentan al menos un DPRQ. Los genes qnr presentes en estos aislados fueron qnrB4 y/o qnrS (algunos aislados también portaban el gen aac(6′)-Ib-cr), los cuales se asocian a mayor incremento de la CMI de fluoroquinolonas3,16. Acorde con nuestros datos, la presencia de aislados con una CMI de fluoroquinolonas elevada en ausencia de mutaciones en gyrA y parC y portadores de DPRQ ha sido descrita recientemente en Polonia2.
La presencia de genes qnr favorece la aparición de mutaciones cromosómicas que reducen la permeabilidad a las fluoroquinolonas o incrementan su expulsión del interior celular (mediante la reducción del número de porinas, las alteraciones en el lipopolisacárido o el incremento de la expresión de bombas de expulsión), lo que determina un incremento de la CMI de ciprofloxacino17,18. Además, las mutaciones que afectan la entrada y la salida de quinolonas aparecen antes que las mutaciones en las regiones QRDR19, algo que concuerda con los datos obtenidos en este estudio, donde se observa que en la mayoría de los aislados con CMI de ciprofloxacino entre 0,5 y 2mg/l no se da la combinación de DPRQ y mutaciones cromosómicas en las regiones QRDR. Por otro lado, la combinación de ambos mecanismos sí se observa en cepas con CMI superior a 2mg/l. Por tanto, la elevada CMI en estos aislados podría deberse a una combinación sinérgica de los DPRQ y mutaciones cromosómicas que reducen la concentración intracelular de quinolonas, de modo que en pasos posteriores estos aislados pudieran adquirir mutaciones en la región QRDR de los genes gyrA y parC, incrementándose el nivel de resistencia a fluoroquinolonas.
Una de las principales limitaciones existentes cuando se estudia la prevalencia de los DPRQ es la ausencia de marcadores fenotípicos de los mismos13, lo que condiciona que la mayoría de los datos de prevalencia de estos genes de resistencia se lleve a cabo en poblaciones concretas: aislados resistentes a fluoroquinolonas o productores de BLEE20-22, lo que dificulta la comparación entre estudios.
En el presente estudio la prevalencia de DPRQ en cepas productoras de betalactamasas de claseC adquiridas y/o carbapenemasas ha sido del 32%, valor similar a los hallados en colecciones parecidas23. Los datos referentes a las cepas productoras de carbapenemasas indican una prevalencia de DPRQ del 39%, valor inferior a los encontrados en otras regiones del mundo, como China, en cepas resistentes a carbapenémicos24. La información obtenida de los aislados productores de carbapenemasas hay que tomarla con cautela debido al bajo número de cepas productoras de estas betalactamasas incluidas en este estudio (28). Además, desde el año 2010 se ha producido una expansión de las carbapenemasas en España, principalmente debido a la dispersión de OXA-48, convirtiéndose esta enzima en la más prevalente en nuestro país25, por lo que sería interesante conocer la prevalencia de DPRQ de los aislados productores de esta betalactamasa en España. A este respecto, se ha descrito recientemente la coexistencia de OXA-48 y AAC(6′)-Ib-cr en K.pneumoniae en Turquía26.
En nuestra colección el DPRQ más prevalente fue qnrB4 (20%), seguido del gen aac(6′)-Ib-cr (7%). Estos datos difieren de lo publicado en multitud de estudios donde el DPRQ más prevalente es aac(6′)-Ib-cr, seguido de qnrB21,24,27. Esta discordancia se debe a la naturaleza de la colección objeto de este estudio, donde la alta prevalencia de qnrB4 se debe a su asociación con DHA-128,29, habiéndose detectado qnrB4 en el 92% de los aislados productores de esta betalactamasa de claseC plasmídica. Un análisis previo en 22 de las cepas que se han considerado para este estudio ya había permitido comprobar que en la gran mayoría de los casos qnrB4 estaba presente en el entorno genético del gen blaDHA-130. Dadas las diferentes poblaciones analizadas en los distintos estudios sobre prevalencia de DPRQ llevados a cabo a nivel mundial resulta difícil la comparación entre ellos. Sería interesante la realización de estudios de prevalencia utilizando poblaciones bacterianas sin sesgos, de modo que podamos obtener datos de prevalencia sin sobre ni subestimaciones. A este respecto, recientemente se ha publicado un algoritmo que permite la detección fenotípica de DPRQ en cepas carentes de modificaciones en QRDR31, lo que facilitaría realizar un estudio de prevalencia de estos mecanismos de resistencia.
Los resultados obtenidos apoyan lo expuesto por otros autores3,16,32, que aunque los DPRQ a quinolonas por sí solos ocasionan un moderado nivel de resistencia a quinolonas, su asociación con mecanismos cromosómicos puede llevar los valores de CMI de las quinolonas por encima de los puntos de corte establecidos, por lo que sería conveniente conocer la epidemiología de estos mecanismos plasmídicos y poder controlar su dispersión, hecho que podría ayudar a reducir o evitar el incremento de la resistencia a quinolonas.
FinanciaciónEste estudio fue parcialmente financiado por el Ministerio de Sanidad, Servicios Sociales e Igualdad, Instituto de Salud Carlos III-FEDER, Red Española de Investigación en Patología Infecciosa (REIPI RD06/0008), por subvenciones del Fondo de Investigación Sanitaria (PS09/00125 y PI11/01117) y por ayudas de AstraZeneca Farmacéutica España y Wyeth (actualmente Pfizer).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
A los miembros de los grupos GEMARA y GEIH de la SEIMC.