La vigilancia epidemiológica de la gripe requiere la recogida de frotis nasofaríngeos en atención primaria para su análisis en laboratorios de referencia. Evaluamos la influencia en el resultado de laboratorio, de los tiempos transcurridos desde el inicio de síntomas hasta la recogida del frotis (TSF) y desde entonces hasta su procesamiento en laboratorio (TFL).
MétodosAnalizamos las muestras recogidas en la red centinela de gripe de Navarra en la temporada 2009-2010. Los hisopados se conservaron refrigerados hasta su estudio mediante RT-PCR y cultivo viral. Se analizó el porcentaje de positividad a gripe en función del TSF y del TFL mediante regresión logística.
ResultadosSe analizaron 937 frotis y 373 (40%) fueron positivos para gripe mediante RT-PCR. El TSF osciló entre 0-15 días. En el análisis ajustado por periodo, laboratorio y edad, la detección del virus de la gripe descendió a menos de la mitad en el cultivo cuando el TSF era de 4-5 días (OR = 0,47; IC 95% 0,24-0,94), y en la RT-PCR, cuando el TSF era mayor de 5 días (OR = 0,24; IC 95% 0,09-0,65). El TFL no afectó de forma significativa al resultado de muestras procesadas por RT-PCR (OR por día transcurrido = 0,96; IC 95% 0,88-1,04), ni por cultivo viral (OR por día transcurrido = 0,97; IC 95% 0,89-1,06).
ConclusionesUn TSF superior a 3 días redujo la probabilidad de confirmación de gripe, afectando más al cultivo que a la PCR. El TFL dentro de un rango de dos semanas no afectó de forma relevante al resultado de la RT-PCR ni del cultivo.
Influenza surveillance requires the collection of nasopharyngeal swabs in Primary Care for testing in reference laboratories. We evaluated the influence on the laboratory results of the time since the onset of symptoms to swabbing (TSS) and from then until laboratory processing (TSL).
MethodsWe analysed swabs collected in the Sentinel Network of Navarra during the 2009-2010 influenza season. The samples were kept refrigerated until analysed by RT-PCR and viral culture. We analysed the percentage of positive swabs to influenza virus in accordance with the TSS and TSL by logistic regression.
ResultsFrom a total of 937 swabs, 373 (40%) were positive for influenza by RT-PCR. The TSS ranged from 0-15 days. In the adjusted analysis by period, laboratory and age, having a positive influenza culture decreased to less than half when the TSS was 4-5 days (OR=0.47; 95% CI, 0.24-0.94), and having a positive RT-PCR decreased when the TSS was 5 days or more (OR=0.24, 95% CI, 0.09-0.65). TSL does not significantly affect the result of the RT-PCR (OR by each day=0.96; 95% CI, 0.88-1.04), or the result of the viral culture (OR by each day=0.97, 95% CI, 0.89-1.06).
ConclusionsA TSS over 3 days reduced the likelihood of confirmation of influenza, affecting the viral culture more than the RT-PCR. A TSL within a range of two weeks had no significant effect on the results of the RT-PCR or the viral culture.
La vigilancia epidemiológica y virológica de la gripe se realiza mediante redes de médicos centinela de atención primaria1,2. La monitorización virológica requiere la recogida de frotis nasofaríngeos en las consultas de atención primaria para su análisis en laboratorios de referencia. Uno de los objetivos de esta actividad es conseguir una buena representatividad geográfica y una cobertura continua durante toda la temporada gripal. El envío diario de las muestras desde lugares dispersos puede ser costoso. Además, las vacaciones y festivos ocasionan ineludiblemente retrasos en los envíos y en el procesamiento de muestras en los laboratorios.
Algunos trabajos habían señalado la progresiva disminución en la detección del virus de la gripe con el transcurso del tiempo desde el inicio de los síntomas3–5, aunque puede haberse modificado con la introducción de técnicas de laboratorio más sensibles, como la reacción en cadena de la polimerasa en transcripción reversa en tiempo real (RT-PCR)6,7 y la aparición del virus A(H1N1)20098.
En este estudio evaluamos la influencia en la detección de gripe en el laboratorio del tiempo transcurrido desde el inicio de los síntomas hasta la recogida del frotis (TSF), y del tiempo entre la toma del frotis y su procesamiento en el laboratorio (TFL).
Material y métodosEn el presente estudio se incluyeron todas las muestras recogidas en la red de médicos centinela de gripe de Navarra durante el periodo de circulación del virus de la gripe A(H1N1)2009 (semanas 22/2009 a 2/2010)9. Dicha red estaba formada por 83 médicos y pediatras de atención primaria previamente formados en el procedimiento de recogida de frotis. Fueron candidatos a la toma de muestra todos los pacientes con síndrome gripal en fase aguda, previo consentimiento verbal. Para mejorar la sensibilidad en la detección de virus, a cada paciente se le tomó doble frotis, faríngeo y nasofaríngeo10. Ambos se introdujeron en un mismo medio de transporte de virus (Viral-Pack, Biomedics), y fueron procesados posteriormente en el laboratorio como una muestra única. Los hisopados se mantuvieron entre 4 y 8° C hasta su envío al laboratorio. Junto con el frotis, el médico recogió datos epidemiológicos, la fecha de inicio de síntomas y la de toma de frotis.
Las muestras fueron procesadas en los dos laboratorios de gripe de Navarra, según la procedencia geográfica. Ambos realizaron RT-PCR de todos los hisopados (Laboratorio A: Influenza A/H1N1 Detection Set, Roche; Laboratorio B: PROFLU Influenza A Subtyping, Prodesse), y en el laboratorio A se realizó además cultivo viral en shell-vial de la línea celular MDCK.
En análisis separados se evaluó el porcentaje de detección de virus de la gripe en los hisopados en función del TSF y del TFL. Ambos análisis se repitieron para las determinaciones realizadas por RT-PCR y por cultivo viral. Mediante regresión logística se evaluaron estas asociaciones ajustando por periodo epidemiológico, la edad del paciente y el laboratorio. El periodo epidemiológico se categorizó en cinco intervalos agrupando semanas consecutivas con porcentajes de detección viral similares.
ResultadosDe los 937 frotis procesados mediante RT-PCR, 373 (40%) resultaron positivos a gripe. En 765 frotis se realizó también cultivo viral y en 284 (37%) se aisló el virus de la gripe. Todas las identificaciones de gripe correspondieron al virus A(H1N1)2009. La edad media de los pacientes fue de 33,5 ± 18,0 años. El porcentaje de identificaciones virales mediante RT-PCR fue mayor en menores de 15 años (59%), en comparación con los de 15 a 59 años (39%) y los mayores de 60 años (18%; p < 0,001), y también varió en función del periodo (entre 8% en las semanas 32 a 40 y 55% en las semanas 41 a 48 de 2009; p < 0,001). No hubo diferencias en el porcentaje de positividad a gripe entre laboratorios (p = 0,935).
En 845 (90%) pacientes se pudo establecer la fecha de inicio de síntomas, y en 339 (40%) de ellos se identificó el virus de la gripe mediante RT-PCR. El TSF osciló entre 0 y 14 días (mediana = 2), si bien, en el 88% de los pacientes el frotis se tomó en los 3 primeros días (tabla 1).
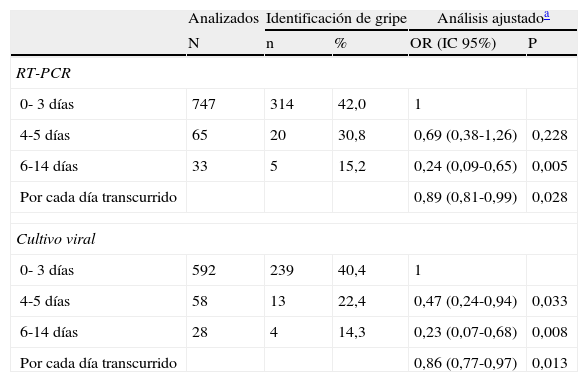
Influencia del tiempo transcurrido entre el inicio de síntomas y la toma de frotis en la probabilidad de identificación del virus de la gripe, según la técnica de laboratorio
| Analizados | Identificación de gripe | Análisis ajustadoa | |||
| N | n | % | OR (IC 95%) | P | |
| RT-PCR | |||||
| 0- 3 días | 747 | 314 | 42,0 | 1 | |
| 4-5 días | 65 | 20 | 30,8 | 0,69 (0,38-1,26) | 0,228 |
| 6-14 días | 33 | 5 | 15,2 | 0,24 (0,09-0,65) | 0,005 |
| Por cada día transcurrido | 0,89 (0,81-0,99) | 0,028 | |||
| Cultivo viral | |||||
| 0- 3 días | 592 | 239 | 40,4 | 1 | |
| 4-5 días | 58 | 13 | 22,4 | 0,47 (0,24-0,94) | 0,033 |
| 6-14 días | 28 | 4 | 14,3 | 0,23 (0,07-0,68) | 0,008 |
| Por cada día transcurrido | 0,86 (0,77-0,97) | 0,013 | |||
IC: intervalo de confianza; OR: odds ratio; RT-PCR: reacción en cadena de la polimerasa en transcripción reversa en tiempo real.
En el análisis de regresión logística, el TSF se asoció a una progresiva reducción en la proporción de identificaciones del virus de la gripe. En comparación con los frotis tomados en los 3 primeros días, los tomados entre 4 y 5 días tuvieron un OR = 0,69 (IC 95% 0,38-1,26), y la caída en la detección viral se hizo más pronunciada a partir del sexto día (OR = 0,24; IC 95% 0,09-0,65) (tabla 1). No se detectó el virus en muestras tomadas 9 días o más después del comienzo de síntomas.
En 678 frotis se realizó además cultivo viral. La proporción de muestras con aislamiento viral también cayó con el transcurso del TSF. En el análisis multivariante, la proporción de aislamientos virales en cultivo se redujo a menos de la mitad cuando habían pasado 4-5 días desde el comienzo de los síntomas (OR = 0,47; IC 95% 0,24-0,94), y a menos de una cuarta parte tras 6 días del inicio de los síntomas (OR = 0,23; IC 95% 0,07-0,68). Introduciendo el TSF en días como variable continua en el modelo multivariante, se observó un descenso en el porcentaje de positividad del 11% por día para la técnica de RT-PCR (OR = 0,89; IC 95% 0,81-0,99) y del 14% para el cultivo viral (OR = 0,86; IC95% 0,77-0,97) (tabla 1).
El TFL se evaluó en los 937 frotis. Osciló entre 0 y 15 días (mediana = 3), pero casi la mitad (49%) de las muestras se procesaron en los 2 primeros días tras haber sido tomadas, y el 75% en los 4 primeros días. El porcentaje de identificaciones virales apenas cambió con el TFL (p = 0,94). Se llegaron a confirmar frotis con TFL de hasta 15 días.
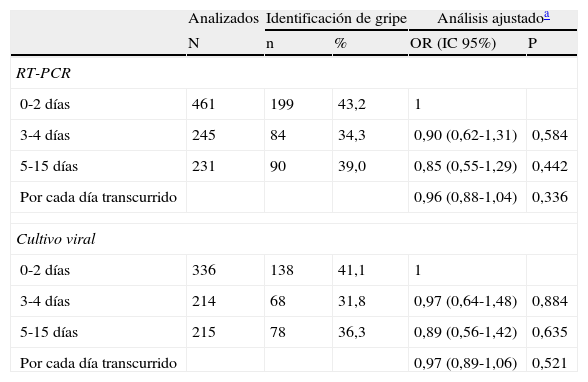
En el análisis de regresión logística, el TFL no afectó de forma significativa al porcentaje de detección de gripe mediante RT-PCR. En comparación con frotis procesados en los dos primeros días, los que tardaron 3 o 4 días presentaron una OR = 0,90 (IC 95% 0,62-1,31), y los que tardaron más de 4 días una OR = 0,85 (IC 95% 0,55-1,29). Considerando el tiempo hasta el procesamiento de la muestra como una variable continua en días en el modelo multivariante se obtuvo una OR = 0,96 (IC 95% 0,88-1,04). En un análisis similar de las muestras que fueron procesadas mediante cultivo viral tampoco se observó que el TFL afectase al porcentaje de identificaciones del virus de la gripe (tabla 2).
Influencia del tiempo transcurrido entre la toma de frotis y su procesamiento en el laboratorio en la probabilidad de identificación del virus de la gripe, según la técnica de laboratorio
| Analizados | Identificación de gripe | Análisis ajustadoa | |||
| N | n | % | OR (IC 95%) | P | |
| RT-PCR | |||||
| 0-2 días | 461 | 199 | 43,2 | 1 | |
| 3-4 días | 245 | 84 | 34,3 | 0,90 (0,62-1,31) | 0,584 |
| 5-15 días | 231 | 90 | 39,0 | 0,85 (0,55-1,29) | 0,442 |
| Por cada día transcurrido | 0,96 (0,88-1,04) | 0,336 | |||
| Cultivo viral | |||||
| 0-2 días | 336 | 138 | 41,1 | 1 | |
| 3-4 días | 214 | 68 | 31,8 | 0,97 (0,64-1,48) | 0,884 |
| 5-15 días | 215 | 78 | 36,3 | 0,89 (0,56-1,42) | 0,635 |
| Por cada día transcurrido | 0,97 (0,89-1,06) | 0,521 | |||
IC: intervalo de confianza; OR: odds ratio; RT-PCR: reacción en cadena de la polimerasa en transcripción reversa en tiempo real.
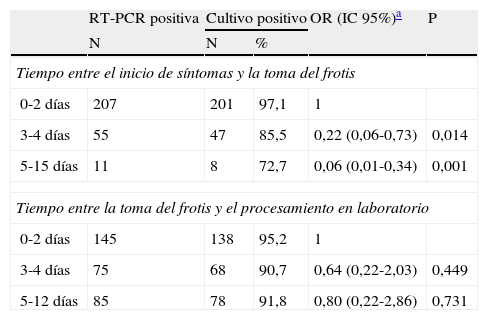
En 273 muestras que se habían confirmado para virus de la gripe por RT-PCR se evaluó el resultado del cultivo viral, consiguiéndose el aislamiento viral en el 94% de ellas. A partir del tercer día de TSF se vio muy reducida la probabilidad de aislamiento del virus de la gripe en el cultivo. Por el contrario, el TFL no afectó significativamente a la probabilidad de aislamiento viral en el cultivo (tabla 3).
Influencia del tiempo transcurrido entre el inicio de síntomas y la toma de frotis y entre éste y el procesamiento de laboratorio en el resultado del cultivo viral entre las muestras con resultado positivo en la reacción en cadena de la polimerasa en transcripción reversa en tiempo real (RT-PCR)
| RT-PCR positiva | Cultivo positivo | OR (IC 95%)a | P | ||
| N | N | % | |||
| Tiempo entre el inicio de síntomas y la toma del frotis | |||||
| 0-2 días | 207 | 201 | 97,1 | 1 | |
| 3-4 días | 55 | 47 | 85,5 | 0,22 (0,06-0,73) | 0,014 |
| 5-15 días | 11 | 8 | 72,7 | 0,06 (0,01-0,34) | 0,001 |
| Tiempo entre la toma del frotis y el procesamiento en laboratorio | |||||
| 0-2 días | 145 | 138 | 95,2 | 1 | |
| 3-4 días | 75 | 68 | 90,7 | 0,64 (0,22-2,03) | 0,449 |
| 5-12 días | 85 | 78 | 91,8 | 0,80 (0,22-2,86) | 0,731 |
IC: intervalo de confianza; OR: odds ratio.
El tiempo entre la toma del frotis y su procesamiento en el laboratorio parece tener poca repercusión en el resultado de la detección de virus de la gripe, dentro de un rango de dos semanas, y siempre que la muestra haya sido conservada adecuadamente. Esto parece ser válido para las determinaciones mediante RT-PCR o mediante cultivo viral. En contraste con ello, el tiempo entre el comienzo de síntomas y la recogida del frotis fue decisivo para poder detectar la presencia de virus. La probabilidad de detectar el virus de la gripe se redujo a menos de la mitad a partir de 6 días del inicio de síntomas para la técnica de RT-PCR, y a partir de 4 días para el cultivo viral. Mientras el cultivo requiere la viabilidad del virus11, la RT-PCR detecta la presencia de genoma, y esto probablemente explica que la RT-PCR tenga un margen de detección más prolongado5, mientras siga habiendo presencia del virus aunque no sea viable3–5.
El cuadro clínico definido como síndrome gripal puede ser producido, no solo por el virus de la gripe, sino también por muchos otros agentes infecciosos: virus parainfluenza, virus respiratorio sincitial, adenovirus, rinovirus y metapneumovirus, entre otros. Estos agentes infecciosos pueden explicar una parte de los casos en los que la detección del virus de la gripe es negativa12. Sin embargo, en nuestro análisis muy probablemente las diferencias en el porcentaje de detección de gripe, en sujetos de una misma edad y en un mismo momento epidemiológico, serán debidas al aumento de resultados falsos negativos.
Entre casos graves de gripe y en inmunodeprimidos la persistencia del virus gripal en nasofaringe puede ser más prolongada13,14. Por tanto, las conclusiones de este estudio no son aplicables a dichas situaciones que se dan con más frecuencia en pacientes de hospitales. En este estudio todas las confirmaciones de gripe correspondieron al virus A(H1N1)20097, por ello, convendría realizar estudios similares en temporadas con otros tipos de gripe, para confirmar si estos hallazgos son generalizables a otros virus.
Estos resultados tienen implicaciones prácticas para la vigilancia de gripe. Los protocolos deberían limitar la toma de frotis a los primeros días tras el comienzo de síntomas, para que la información obtenida sea válida. Los frotis negativos tomados después del cuarto día tras inicio de síntomas no descartan que el paciente haya tenido infección por virus de la gripe. En esta situación la RT-PCR podría proporcionar resultados más válidos que el cultivo. El tiempo para el envío de frotis al laboratorio no afecta tanto al resultado, y muestras que lleguen con varios días de demora no tiene por qué aportar resultados inválidos, si se han conservado refrigeradas.
En conclusión, encontramos que el tiempo desde la toma del frotis hasta su procesamiento en el laboratorio, dentro de un rango de dos semanas, afecta poco a los resultados de identificación del virus de la gripe, mientras que el tiempo entre el inicio de los síntomas y la toma del frotis conlleva un aumento de resultados falsos negativos a partir del cuarto día, viéndose más afectado el cultivo viral que la RT-PCR.
FinanciaciónInstituto de Salud Carlos III (GR09/0028 y PS09/01179). Proyecto I-MOVE financiado por el Centro Europeo para el Control de Enfermedades (ECDC).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Red de Médicos Centinela de Gripe de Navarra 2009-2010: I. Abad, J. Agreda, P. Aldaz, E. Álvarez, J.J. Arana, I. Arceiz, E. Arina, M.D. Artajo, A. Arza, K. Ayerdi, B. Azagra, J. Baleztena, J. Bartolomé, C. Bernués, J. Berraondo, C. Bolea, A. Brugos, S. Buil, F. Calle, B. Cano, J.C. Cenoz, F. Cía, F. Cortés, C. Chérrez, B. Churío, E.M. Da Costa, J. De Prado, J. Díez Espino, M. Doiz, F.J. Escribano, M.J. Esparza, L. Fanlo, C. Fernández Alfaro, A. Fernández Urtasun, J. Gamboa, M.L. Garcés, P. González Lorente, N. Goñi, J. Guillén, M.J. Guillorme, J.O. Guiu, A. Gulina del Pueyo, J.C. Gurbindo, M.J. Guruchaga, J.A. Heras, M.S. Indurain, B. Iñigo, S.E. Juan Belloc, O. Lecea, M.P. León, J.J. Longás, A. Martínez Díaz, C. Maurer, M. Monge, M. Moreno, U. Navarro, F.J. Orozco, M. Orte, J. Palau, F. Pérez Afonso, M.L. Pérez Del Valle, P. Pérez Pascual, M.A. Pous, A. Prado, A. Puig Arrastia, E. Ridruejo, M.A. Rodríguez González, A. Roig, M.A. Roncal, I. Ruiz Puertas, H. Selles, M.A. Senosiain, J. Sola, M. Sota, P. Uhalte, J. Ulibarri, M.E. Ursua, I.A. Urtasun, M.J. Vigata, M.T. Virto, J.M. Vizcay, M. Zabalza, J. Zubicoa.
El listado de los miembros de la Red de Médicos Centinela de Gripe de Navarra se presenta en el Anexo 1.