La resistencia de los microorganismos de aislamiento clínico frecuente requiere, por parte del laboratorio de microbiología, de la realización de pruebas que permitan la detección fenotípica de los mecanismos subyacentes responsables de los perfiles observados in vitro. La comunicación de estos fenotipos, ya sea inferidos o demostrados, permite adecuar el tratamiento antibiótico y reunir información para el seguimiento epidemiológico de las resistencias. En este trabajo se detallan las diferentes pruebas que se realizan para la detección fenotípica de la resistencia a las principales familias de antimicrobianos utilizados frente a los microorganismos grampositivos de mayor incidencia clínica como son Staphylococcus spp., Enterococcus spp. y Streptococcus pneumoniae. En Staphylococcus, se describen las pruebas fenotípicas para evidenciar los mecanismos de resistencia a betalactámicos así como a los macrólidos, lincosamidas y estreptograminas B (MLSB), la resistencia disminuida a glucopéptidos, a aminoglucósidos, a linezolid y a mupirocina. En Enterococcus se detallan las pruebas para detectar los fenotipos de resistencia a glucopéptidos y la resistencia de alto nivel a los aminoglucósidos. En S. pneumoniae, se destaca la importancia de la detección de resistencia a la penicilina y la sensibilidad disminuida a cefalosporinas de 3.a generación así como la sensiblidad disminuida o la resistencia a fluoroquinolonas.
Antimicrobial resistance mechanisms among clinically relevant gram-positive microorganisms can be demonstrated using phenotypic tests that enable the interpretation of underlying mechanisms responsible for the in vitro resistance. The reporting of these mechanisms, either inferred or demonstrated, helps in the adjustment of clinical treatments and the epidemiological follow up of resistance traits. In the present work, phenotypic tests for detection of antimicrobial resistance mechanisms involving the most frequent antimicrobial families used against Staphylococcus spp., Enterococcus spp. and Streptococcus pneumoniae are analysed. In the case of Staphylococcus, phenotypic tests to reveal the mechanisms of resistance against beta-lactams, macrolides, lincosamides and streptogramin B (MLSB), as well as intermediate susceptibility to glycopeptides, resistance to aminoglycosides, mupirocin and linezolid are reviewed. Tests to detect glycopeptide resistance and high-level aminoglycoside resistance among enterococci are analysed. Detection of penicillin resistance, as well as diminished susceptibility to third generation cephalosporins, together with diminished susceptibility or even resistance to fluoroquinolones is also detailed.
El impacto clínico de la resistencia antimicrobiana requiere el estudio de los mecanismos implicados con el fin de contribuir a una adecuación rápida y dirigida del tratamiento así como para el seguimiento y el control epidemiológicos.
En la presente revisión se detallan las pruebas in vitro para la detección e inferencia fenotípica de los mecanismos de resistencia subyacentes más frecuentes en los géneros Staphylococcus, Enterococcus y en Streptococcus pneumoniae frente a las familias de antimicrobianos de uso habitual. En el caso de los estafilococos se describen las pruebas para la detección de resistencia a los betalactámicos, en particular a la oxacilina en sus distintas formas, homogénea, heterogénea y borderline; los mecanismos implicados en la resistencia a MLSB (macrólidos, lincosamidas y estreptograminas B); los que afectan a los aminoglucósidos; al linezolid y a la mupirocina así como los responsables de la sensibilidad disminuida a los glucopéptidos. En los enterococos se abordan la resistencia de alto nivel a los aminoglucósidos (gentamicina y estreptomicina) y la resistencia a los glucopéptidos. Finalmente, en el caso de neumococo, se detalla la resistencia a los betalactámicos y las diferentes PBP implicadas en cada caso con referencia explícita a la interpretación de los distintos niveles de sensibilidad según se trate de aislados meníngeos o no meníngeos. Asimismo, en esta especie se analiza la resistencia a las fluoroquinolonas y el fundamento del desarrollo escalonado de la misma así como las pruebas para la detección del primer o bajo nivel de resistencia a estos compuestos. Los puntos de corte considerados para la interpretación de la sensibilidad son los indicados por el Clinical and Laboratory Standards Institute (CLSI, 2011) si bien en determinados casos se incluyen los correspondientes al European Committee on Antimicrobial Susceptibility Testing (EUCAST, 2011) y al Comité de l‘Antibiogramme de la Société Française de Microbiologie (SFM, 2010)1–3.
Resistencia a los antimicrobianos en estafilococosResistencia a los betalactámicosResistencia a las penicilinas por producción de betalactamasaEl fenotipo de resistencia a la penicilina mediado por betalactamasa(s) en estafilococo implica resistencia a todas las penicilinas excepto a las isoxazolilpenicilinas: oxacilina, meticilina, cloxacilina y nafcilina así como sensibilidad a las combinaciones de betalactámico con inhibidor de betalactamasa (ácido clavulánico, tazobactam y sulbactam), a las cefalosporinas y a las carbapenemas.
Las penicilinasas estafilocócicas son betalactamasas de clase A, por tanto, sensibles a los inhibidores clásicos ya mencionados y de las que se han descrito cuatro tipos: A, B, C y D, si bien la más frecuente es la de tipo C4. Algunas de estas penicilinasas pueden hidrolizar ciertas cefalosporinas en presencia de inóculos elevados (efecto de inóculo). Así por ejemplo, existe un marcado efecto de inóculo sobre la cefalexina si el estafilococo produce las betalactamasas de tipo A o C y un efecto de inóculo moderado sobre la cefalotina si produce las de tipo B o C. En ningún caso existe efecto de inóculo frente a cefuroxima ni frente a ceftriaxona5.
Detección fenotípica de resistencia por producción de betalactamasasPara la detección de cepas de Staphylococcus aureus resistentes a la penicilina por producción de betalactamasa es más adecuado utilizar un disco de 10 unidades de penicilina que un disco de 10μg de ampicilina. El resultado obtenido con este disco de penicilina se debe extrapolar a todas las penicilinas lábiles a la acción de la penicilinasa, como ampicilina, amoxicilina, carbenicilina, ticarcilina, azlocilina, mezlocilina y piperacilina. Para la determinación de la CMI a las penicilinas se debe utilizar el medio de Mueller-Hinton, cuya concentración de cationes (Ca+2 y Mg+2) debe estar ajustada en el caso de que se realice mediante el método de dilución en caldo; se requiere un inóculo equivalente al 0,5 de la escala de McFarland y entre 18-20 horas de incubación a 35-37°C. Cuando el halo de inhibición con el disco de penicilina de 10 unidades sea de≥29mm (sensible) o por dilución en caldo o en agar, la CMI de penicilina sea de≤0,12mg/L (sensible) se debe confirmar que el microorganismo es realmente sensible a las penicilinas; para ello, se realiza una prueba con la cefalosporina cromogénica nitrocefin utilizando las colonias crecidas en el borde del halo de inhibición de la penicilina ya que en estas, la producción de penicilinasa ha sido inducida por la incubación previa en presencia del antimicrobiano. Esta prueba consiste en depositar con el asa parte de esas colonias sobre un disco impregnado con el nitrocefin que al hidrolizarse en presencia de la betalactamasa experimenta un cambio en su estructura dando un producto coloreado (naranja-rojo)6.
Resistencia a la meticilina: homogénea y heterogéneaLa resistencia a la meticilina, más frecuente entre las diferentes especies de estafilococos coagulasa negativa (ECN, con la excepción de Staphylococcus lugdunensis y Staphylococcus saprophyticus) que en S. aureus, se debe a la adquisición del gen mecA que codifica la proteína fijadora de penicilina (PBP) PBP2a, supernumeraria, que posee baja afinidad por todos los betalactámicos y por tanto implica resistencia a todos estos compuestos con la excepción, hasta el presente, de la cefalosporina ceftarolina y de la carbapenema razupenem, ninguna de las cuales ha sido aún introducida para uso clínico7,8.
En España, la prevalencia de resistencia a la meticilina en S. aureus (SARM) se mantiene en torno al 30%, mientras que entre las diferentes especies de ECN (ECNRM) las cifras oscilan entre el 60-70%9.
S. aureus posee cuatro PBP de las cuales la 1, 2 y 3 son esenciales. La PBP de baja afinidad denominada PBP2a o PBP2′, de 78kDa, está codificada por el gen cromosómico mecA. La expresión de la resistencia mediada por este gen es compleja y se afecta por diferentes factores como la temperatura, el pH, la osmolaridad así como por la presencia de secuencias cromosómicas reguladoras y de otros genes cromosómicos no relacionados. Esta expresión, tanto en SARM como ECNRM, puede ser homogénea o heterogénea. En las cepas que presentan resistencia homogénea o de alto nivel a la oxacilina, la mayor parte de la población expresa dicha resistencia. Las cepas con expresión heterogénea (CMI de oxacilina 1-16mg/L) se caracterizan porque solo una pequeña proporción de la población (≤0,1%) sobrevive a concentraciones de oxacilina superiores a 10mg/L, mientras que la mayor parte no es viable a bajas concentraciones del antimicrobiano (1-5mg/L). La mayoría de los aislamientos clínicos presentan este patrón de heterorresistencia bajo las condiciones rutinarias de cultivo. Sin embargo, las cepas heterogéneas pueden aparecer como homogéneas bajo ciertas condiciones, como el crecimiento en un medio hipertónico (con 2% de NaCl) o con una incubación a 30°C. Estos cambios en la expresión de la resistencia bajo diferentes condiciones de cultivo son transitorios y solo de expresión fenotípica6,10.
Detección fenotípica de resistencia a la oxacilinaLa resistencia a la meticilina en Staphylococcus se puede detectar en el laboratorio mediante la técnica de difusión con discos de oxacilina (1μg) y/o cefoxitina (30μg) o por dilución en caldo o en agar. Para ello se utiliza el medio de Mueller Hinton con el agregado de 2% de NaCl en el caso de realizar los métodos de dilución y además ajustado con cationes (Ca+2 y Mg+2) en el caso de que se realice mediante el método de dilución en caldo. El inóculo empleado es el equivalente al 0,5 de la escala de McFarland y se requieren 24 horas completas de incubación en atmósfera aerobia a 35°C. La incubación a temperaturas superiores a 35°C puede impedir la detección de esta resistencia. Una cepa de S. aureus se considera resistente a la oxacilina cuando el halo de inhibición de la oxacilina es≤10mm o cuando la CMI de oxacilina es≥4mg/L. En el caso de los ECN, una cepa se considera resistente a la oxacilina cuando la CMI es≥0,5mg/L, excepto en S. lugdunensis que se considera resistente si la CMI de oxacilina es≥4mg/L10,6.
La cefoxitina es un marcador adecuado de la presencia de mecA ya que es un compuesto inductor más potente del sistema regulatorio de mecA que las penicilinas y por ello, al mejorar la expresión de este gen se mejora también la detección de la resistencia a la meticilina. La utilización del disco de cefoxitina es especialmente útil y de preferencia sobre el disco de oxacilina para detectar la resistencia a oxacilina mediada por el gen mecA en las cepas heterorresistentes y se debe utilizar siempre en cepas de ECN. Además, este disco no presenta problemas de estabilidad como la oxacilina durante su conservación.
Las cepas con heterorresistencia suelen aparecer como sensibles a muchos betalactámicos cuando se realiza un antibiograma con discos y su interpretación como tal puede conducir a fracasos terapéuticos. Por tanto, las cepas de estafilococos resistentes a la cefoxitina (halo de ≤21mm en S. aureus, CMI de ≥8mg/L y de ≤24mm en ECN, CMI no definida) indican la presencia de mecA y en consecuencia, la resistencia a todos los betalactámicos. Por el contrario, la sensibilidad a cefoxitina en S. aureus (halo de≥22mm, CMI de ≤4mg/L) o en ECN (halo≥25mm, CMI no definida) descarta la presencia del gen mecA y por tanto la heterorresistencia a la meticilina1,10,11.
Resistencia borderline (límite) a la oxacilina (BORSA) en Staphylococcus aureusExisten cepas de S. aureus que presentan resistencia de bajo nivel o borderline (en el límite) a la oxacilina (borderline oxacillin-resistant Staphylococcus aureus, BORSA) y se caracterizan por tener CMI de oxacilina en el punto de corte de resistencia (4mg/L) o una dilución por encima de este. Estos aislados se dividen en dos grupos en función de la presencia o ausencia del gen mecA: si poseen el gen mecA son cepas heterorresistentes que producen la PBP2a. Si no contienen el gen mecA ni por tanto la PBP2a, esta resistencia de bajo nivel puede deberse a la hiperproducción de la betalactamasa estafilocócica o a la modificación (hiperproducción o alteración) de las PBP 1, 2, y 4. Las cepas que hiperproducen betalactamasa son sensibles a las asociaciones de betalactámico con inhibidor de betalactamasa (ácido clavulánico, tazobactam y sulbactam)12.
Detección fenotípica de resistencia tipo BORSALas cepas BORSA mecA negativas son sensibles a la cefoxitina (halo≥22mm) mientras que las mecA positivas son resistentes a este antimicrobiano (halo≤21mm). Las cepas con resistencia intermedia a la oxacilina pero sensibles a la cefoxitina se deben informar como sensibles a la cefoxitina y nunca se debe informar una sensibilidad intermedia a la oxacilina. Del mismo modo, si se realiza la detección del gen mecA y/o de la PBP2a por técnicas moleculares y por técnicas de aglutinación con látex, respectivamente y los resultados son negativos, el aislamiento debe informarse como sensible a la oxacilina. Estas cepas borderline mecA negativas son sensibles a todos los betalactámicos (penicilinas antiestafilocócicas, cefalosporinas y carbapenemas). Por el contrario, las cepas mecA positivas son resistentes a todos los betalactámicos13. No existen datos clínicos que sugieran que el nivel de resistencia expresado por las cepas borderline mecA negativas conduzca al fracaso terapéutico y los datos obtenidos en estudios en animales demuestran que la oxacilina es eficaz en el tratamiento de infecciones causadas por este tipo de cepas13.
Resistencia a los macrólidos y a la clindamicinaLos macrólidos, las lincosamidas y las estreptograminas B (MLSB) son tres familias diferentes de antimicrobianos que poseen mecanismos y sitios de acción (subunidad 50S del ribosoma bacteriano) similares. En los estafilococos, los fenotipos de resistencia que se pueden observar son: 1) fenotipo cMLSB (resistencia constitutiva a la eritromicina (y demás macrólidos de 14 y 15 átomos de carbono), a la clindamicina y a las estreptograminas B por modificaciones en la diana ARNr 23S debidas a la acción de metilasas codificadas por los genes ermA, ermB, ermC, con resistencia cruzada de alto nivel a todos los antimicrobianos del grupo MLSB; 2) fenotipo iMLSB (resistencia inducible a la eritromicina y sensibilidad a la clindamicina y a las estreptograminas B): se trata del mismo mecanismo que en el caso anterior pero que se manifiesta como resistencia a los macrólidos pero con sensibilidad a las lincosamidas y a las estreptograminas B en ausencia de un inductor como la propia eritromicina, y 3) fenotipo MSB, resistencia a la eritromicina, a otros macrólidos de 14 y de 15 átomos de carbono, a las estreptograminas B pero con sensibilidad a clindamicina, mediada por una bomba de expulsión activa (codificada por genes plasmídicos del tipo msrA). Existe un cuarto fenotipo de resistencia a la clindamicina con sensibilidad a la eritromicina y a la estreptogramina B, poco frecuente, debido a la acción de enzimas (codificadas por los genes lnu) que inactivan exclusivamente a las lincosamidas14.
Detección fenotípica de resistencia a los macrólidos, las lincosamidas y las estreptograminas BLas diferentes expresiones fenotípicas de estas resistencias se pueden identificar en el laboratorio mediante el método de difusión con discos de eritromicina y clindamicina empleando el llamado D-test y por dilución en caldo utilizando una combinación de ambos antimicrobianos6. Para la realización del D-test, se colocan un disco de eritromicina de 15μg y otro de clindamicina de 2μg separados a una distancia de 15 o 20mm sobre la superficie de la placa inoculada, siendo posibles los siguientes resultados: 1) resistencia absoluta a la eritromicina y a la clindamicina sin achatamiento del halo de inhibición de la clindamicina (D-test negativo, cMLSB); 2) resistencia a la eritromicina y sensibilidad a la clindamicina pero con un achatamiento del halo del disco de la clindamicina en la proximidad del de la eritromicina (D-test positivo, iMLSB). En este último fenotipo, si se estudia la sensibilidad a los macrólidos de 16 átomos de carbono, a la clindamicina y a las estreptograminas del grupo B en ausencia de eritromicina (inductor), las cepas aparecerán como sensibles a estos antimicrobianos, cuando en realidad son resistentes ya que poseen el mecanismo que se puede inducir in vivo y conducir a fracasos terapéuticos. Este fenotipo también se puede detectar por el método de microdilución en caldo utilizando la combinación, en el mismo pocillo, de eritromicina (4mg/L) y clindamicina (0,5mg/L), además de pocillos individuales para determinar la CMI de la eritromicina y de la clindamicina por separado. Tras la incubación (35-37°C durante 18-24 horas) una CMI de eritromicina en el rango de resistente junto con una CMI de clindamicina en el rango de sensible, asociados a la presencia de crecimiento en el pocillo combinado con eritromicina y clindamicina, indicarán que se trata del fenotipo de resistencia inducible15,16, y 3) resistencia a la eritromicina (y a los demás macrólidos de 14 y 15 átomos de carbono así como a las estreptograminas B) y sensibilidad a la clindamicina sin achatamiento del halo del disco de esta última (D-test negativo, MSB). Los fenotipos de resistencia derivados de la inactivación de los antimicrobianos de estas tres familias son poco frecuentes. Se han descrito enzimas con actividad fosfotransferasa que inactivan a los macrólidos de 14 y de 15 átomos de carbono y nucleotidiltransferasas que inactivan exclusivamente las lincosamidas (enzimas Lnu).
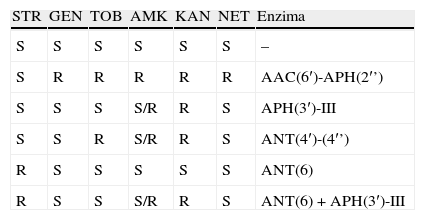
Resistencia a los aminoglucósidosEn la mayoría de los casos se estudia la sensibilidad in vitro de los estafilococos a la gentamicina, a la tobramicina y a la amicacina puesto que estos son los de mayor utilización clínica. Los dos fenotipos más frecuentes son el de resistencia a la gentamicina y a la tobramicina con sensibilidad aparente a la amicacina y el de sensibilidad a la gentamicina y resistencia a la tobramicina y a la amicacina. Las enzimas implicadas en cada caso y los fenotipos que se pueden observar se resumen en la tabla 1.
Fenotipos y enzimas inactivadoras de aminoglucósidos en el género Staphylococcus17
| STR | GEN | TOB | AMK | KAN | NET | Enzima |
| S | S | S | S | S | S | – |
| S | R | R | R | R | R | AAC(6′)-APH(2′’) |
| S | S | S | S/R | R | S | APH(3′)-III |
| S | S | R | S/R | R | S | ANT(4′)-(4′’) |
| R | S | S | S | S | S | ANT(6) |
| R | S | S | S/R | R | S | ANT(6)+APH(3′)-III |
Cuando se requiere la confirmación de un fenotipo de resistencia a aminoglucósidos, esta se realiza por difusión con discos. Habitualmente se emplean discos de gentamicina, tobramicina, amicacina, kanamicina, netilmicina y estreptomicina.
La resistencia a gentamicina debe asociarse con resistencia a todos los aminoglucósidos de utilización clínica (la propia gentamicina, tobramicina, amicacina, netilmicina y además kanamicina, con la excepción de la estreptomicina) y es debida a la actividad de la enzima bifuncional AAC(6′)-APH(2”) (tabla 1). Es importante destacar que la situación opuesta no existe, es decir, no se debe asumir que la sensibilidad a la gentamicina implica también sensibilidad al resto de los aminoglucósidos puesto que la presencia de la enzima ANT(4′)(4”) que determina resistencia a la tobramicina, la kanamicina y sensibilidad variable a la amicacina es frecuente entre las cepas de SARM de los hospitales españoles y del resto de Europa (tabla 1). La tobramicina es el mejor marcador de este mecanismo de resistencia, de modo que aquellas cepas que aparezcan en el antibiograma como sensibles a la gentamicina y resistentes a la tobramicina deben informarse como resistentes también a la amicacina ya que la capacidad bactericida de esta se encuentra disminuida6,17.
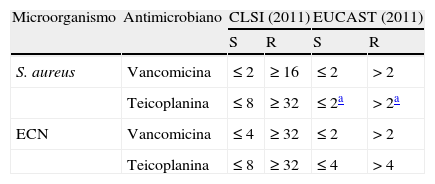
Sensibilidad disminuida a los glucopéptidosEn S. aureus, una cepa se define como vancomycin intermediate S. aureus (VISA) cuando la CMI de vancomicina, determinada por el método de microdilución en caldo, es de 4-8mg/L según los criterios del Clinical and Laboratory Standards Institute (CLSI). Por lo tanto, se considera que una cepa es resistente si la CMI de vancomicina por dicho método es de≥16mg/L. Las cepas VISA pueden presentar también sensibilidad disminuida o resistencia a la teicoplanina y se las denomina glycopeptide-intermediate S. aureus (GISA). En el caso de ECN se considera que una cepa tiene sensibilidad intermedia a la vancomicina (CLSI) cuando los valores de CMI son de 8-16mg/L y por tanto que es resistente cuando la CMI es≥32mg/L. Los criterios de sensibilidad de CLSI y de EUCAST para los glucopéptidos en el género Staphylococcus se muestran en la tabla 2. Además, existe también el fenómeno de heterorresistencia a los glucopéptidos, aunque es menos frecuente que la sensibilidad intermedia y se ha observado generalmente después de un tratamiento prolongado con glucopéptidos. Una cepa de S. aureus heterorresistente (hVISA) es aquella frente a la cual la CMI de vancomicina, determinada por microdilución, está en el rango de sensible (CMI≤2mg/L) pero para la que la CMI de este antibiótico frente a una proporción de las células de su población está en el rango intermedio (4-8mg/L)18.
Puntos de corte (CMI, mg/L) definidos por CLSI y por EUCAST para vancomicina y teicoplanina frente a Staphylococcus1,2
La determinación de la sensibilidad a vancomicina (y a teicoplanina) por difusión con discos está desaconsejada por su falta de capacidad discriminatoria tanto para S. aureus como para ECN y por tanto no se han definido puntos de corte1,2.
En las cepas con expresión heterogénea, la población resistente está presente con una frecuencia de≤10−5 a 10−6, su detección resulta muy difícil con los métodos estándar de microdilución y de dilución en agar en los que se utiliza un inóculo aproximado de 5×104UFC por pocillo o de 1×104UFC por depósito, respectivamente. Por otra parte, las cepas con fenotipo de sensibilidad intermedia tienen una escasa estabilidad en ausencia de glucopéptidos, de manera que con frecuencia, revierten al fenotipo sensible tras sucesivos subcultivos en medios que no contienen estos compuestos6,18.
Se han evaluado varios métodos para el cribado y la confirmación de las cepas de estafilococos con resistencia intermedia o con heterorresistencia a los glucopéptidos. En algunas ocasiones sus características morfológicas son diferentes a las de las cepas sensibles con apariencia de cultivos mixtos de colonias grandes y pequeñas de pigmentación variable. Se debería determinar la sensibilidad a la vancomicina de cada uno de los diferentes morfotipos, principalmente en el caso de S. aureus. Para detectar estas cepas con sensibilidad disminuida a la vancomicina se debe realizar la CMI por dilución en caldo o mediante Etest o bien mediante la inoculación de una placa de agar Brain Heart Infusion (BHI) de cribado con 6mg/L de vancomicina y con una incubación de 24 horas1. Hay que tener en cuenta que las CMI obtenidas mediante Etest suelen ser siempre una dilución superior a las obtenidas por microdilución y que el método que define a una cepa como VISA o hVISA es el de microdilución. Además y debido a las diferencias en los resultados de CMI según el método por el que esta se determina, en el informe debe constar el método empleado para contribuir a la toma de decisiones terapéuticas. Otra alternativa para su detección consiste en utilizar el denominado macrométodo en el que se determina la CMI mediante Etest pero con un inóculo equivalente al 2 de la escala de McFarland. Este método constituye solamente una herramienta de cribado para detectar estas cepas pero el resultado obtenido no es la verdadera CMI, y por tanto, no se debe informar como tal. No obstante, la detección precisa de este fenotipo requiere un análisis poblacional (population analysis profile, PAP). Para realizar este análisis la cepa patrón a utilizar es la cepa hVISA Mu3 (S. aureus ATCC 700698). Utilizando el PAP como método de referencia se puede detectar el fenotipo hVISA en cepas de S. aureus con CMI de vancomicina tan bajas como 0,5 o 1mg/L6,17,18.
Resistencia a la mupirocinaLa mupirocina o ácido pseudomónico es un antimicrobiano de uso exclusivamente tópico que se utiliza para la descolonización de portadores nasales de S. aureus. Se pueden observar dos fenotipos de resistencia a la mupirocina: 1) resistencia de bajo nivel (mutaciones en el gen cromosómico ileS, que codifica la síntesis de la enzima isoleucil t-ARN sintetasa de baja afinidad, con CMI entre 8 y 256mg/ml), y 2) alto nivel de resistencia (adquisición de un gen plasmídico, ileS-2 o mupA que codifica una isoleucil t-ARN sintetasa sin afinidad por la mupirocina, CMI≥512mg/ml)19,20.
Detección fenotípica de la resistencia a la mupirocinaAunque el CLSI no ha definido puntos de corte para interpretar la sensibilidad a la mupirocina, en su último documento establece las técnicas para la detección de cepas con resistencia a este compuesto1. Para ello indica el uso del disco de 200μg y/o el empleo de un pocillo que contenga 256mg/L de mupirocina si se realiza microdilución (condiciones estándar de temperatura y tiempo de incubación). Cualquier tamaño de halo o falta de crecimiento en el pocillo indican ausencia de alto nivel de resistencia. El EUCAST plantea el uso del disco de 200μg y los puntos de corte de 30mm (S) y de 18mm (R), pero exclusivamente para aquellas cepas implicadas en procesos de colonización nasal que requieran descontaminación2. Asimismo, diferentes estudios recomiendan que se consideren sensibles a aquellas cepas cuya CMI es≤4mg/L o que presentan un halo de inhibición de≥14mm (utilizando discos de 5μg). En el caso de que mediante la técnica de difusión con el disco de 5μg la cepa sea resistente (halo <14mm) será necesario determinar siempre la CMI mediante el método de microdilución o mediante Etest ya que un valor de CMI de 8 o de 256mg/L tiene diferentes implicaciones clínicas. Si no se dispone de estas técnicas se puede utilizar la combinación de discos de mupirocina de 5μg y de 200μg o bien utilizar solamente el de 20μg para determinar si la resistencia es de bajo o de alto nivel21 (tabla 3).
Fenotipos de resistencia a la mupirocina obtenidos mediante el método de difusión con discos y su relación con los valores de CMI (mg/L)19,21
| Sensible (mm halo) | Resistencia de bajo nivel (halo en mm) | Resistencia de alto nivel (halo en mm) | |
| Un disco: 20μg | ≥17 | 6-16 | 0 |
| Dos discos: 5μg200μga | ≥14>6 | <14>6 | <140 |
| CMI(mg/L) | CMI(mg/L) | CMI(mg/L) | |
| ≤4 | 8-256 | ≥512 |
La resistencia al linezolid es actualmente poco frecuente y generalmente se observa en cepas de pacientes con tratamientos prolongados con este antimicrobiano, en el caso de diseminación de clones resistentes en unidades hospitalarias o de diseminación de transposones o plásmidos que contienen el gen cfr.
En el laboratorio se pueden observar tres fenotipos de resistencia al linezolid en estafilococos: 1) resistencia cruzada al linezolid y a las pleuromutilinas debido a mutaciones en el dominio V del ARNr 23S (genes rrn) en los que los niveles de resistencia aumentan en función del número de copias del gen ARNr 23S afectadas; 2) resistencia cruzada al linezolid, los fenicoles, las pleuromutilinas, las lincosamidas y las estreptograminas A por adquisición del gen plasmídico cfr que codifica la producción de una metilasa ribosómica, y 3) resistencia cruzada al linezolid, los macrólidos y el cloranfenicol debida a mutaciones en los genes que codifican las proteínas L3 y L4 de la subunidad ribosómica 50S, codificadas por los genes rplC y rplD, respectivamente. Puede ocurrir que en la misma cepa se den combinaciones simultáneas de estos mecanismos22,23.
Detección fenotípica de la resistencia al linezolidEl método más adecuado para detectar la resistencia al linezolid es la microdilución en caldo. Los resultados de CMI obtenidos por Etest son generalmente más bajos, principalmente cuando la resistencia está mediada por el gen cfr y las CMI pueden ser tan bajas como 1 o 2mg/L (rango sensible). El método de difusión con discos no detecta adecuadamente la resistencia al linezolid, excepto en los casos con alto nivel de resistencia (CMI>32mg/L)1,6,17.
En el caso de las cepas resistentes al linezolid debido a la expresión del gen cfr es característica la resistencia simultánea a la clindamicina y al cloranfenicol con sensibilidad a los macrólidos. Sin embargo, la relativa frecuencia de cepas que poseen dos o tres mecanismos de resistencia diferentes (resistencia al cloranfenicol, resistencia de tipo MLSB, etc.) dificulta la adecuada detección de resistencia al linezolid mediada por este mecanismo. Por su parte, la utilización de discos de quinupristina/dalfopristina no contribuye a su detección puesto que al incluir una estreptogramina A y una estreptogramina B, muchas cepas con el gen cfr pueden ser sensibles a la estreptogramina B y por tanto, sensibles a la combinación. Cuando la resistencia está mediada por alteraciones en las proteínas L3 y L4, se produce resistencia cruzada al cloranfenicol y a los macrólidos, pero como ya se indicó anteriormente, debido a la relativa frecuencia de cepas que presentan mecanismos de resistencia simultáneos que pueden afectar a la actividad de estos compuestos, la presencia de este mecanismo es difícil de inferir6,17,22.
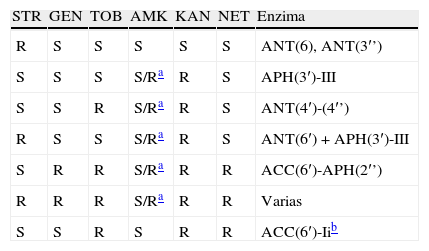
Resistencia a los antimicrobianos en enterococosResistencia de alto nivel a la gentamicina y a la estreptomicinaLos enterococos poseen resistencia intrínseca de bajo nivel a los aminoglucósidos, con CMI entre 4-64mg/L para la gentamicina y 16-256mg/L para la estreptomicina. La adquisición de determinantes genéticos asociados a la producción de enzimas modificadoras de aminoglucósidos conlleva la expresión de altos niveles de resistencia (RAN) a dichos compuestos24. En la tabla 4 se detallan los distintos fenotipos de RAN de origen enzimático así como las combinaciones de enzimas que dan lugar a los distintos fenotipos.
Fenotipos y enzimas modificadoras de aminoglucósidos más frecuentes en el género Enterococcus17,24
La detección de la RAN a la gentamicina y a la estreptomicina en enterococo se puede realizar mediante las siguientes técnicas: 1) difusión con discos de alta carga (gentamicina, 120μg; estreptomicina, 300μg) en agar Mueller Hinton, con un inóculo equivalente al 0,5 de la escala de McFarland e incubación a 35°C durante 16-18h1. En función del halo de inhibición se establece si la cepa presenta RAN a los aminoglucósidos estudiados y con ello si es factible la sinergia de dichos compuestos con antimicrobianos que actúan a nivel de la pared celular. Si el halo de inhibición es≤6mm (tanto para gentamicina como para estreptomicina) se considera que hay RAN y por tanto que no existirá sinergismo. En ocasiones, los resultados de la difusión con disco son poco concluyentes (halos de inhibición en el rango 7-9mm) lo cual requiere la confirmación mediante los siguientes métodos; 2) microdilución en caldo o dilución en agar: se emplea el medio BHI al que se añaden 500mg/L de gentamicina o bien 1.000 o 2.000mg/L de estreptomicina (microdilución en caldo o dilución en agar, respectivamente); tras una incubación durante 24 horas a 35° C cualquier tipo de crecimiento será indicativo de resistencia y por tanto de ausencia de posible sinergia con agentes activos sobre la pared celular6,17; 3) curvas de letalidad: permiten determinar la probabilidad de sinergia entre betalactámicos o glucopéptidos y aminoglucósidos. Es un método laborioso que no se realiza de rutina.
Los métodos aceptados por el CLSI para la detección de RAN a la gentamicina pueden no detectar ciertas cepas portadoras de la enzima APH(2”)-Ic (infrecuente) que confiere bajos niveles de resistencia (CMI 256mg/L), sin embargo, estas cepas se pueden detectar si se utiliza el punto de corte de resistencia >128mg/ml establecido por EUCAST1,2. Esta situación plantea la posibilidad de que en el futuro haya que revisar los puntos de corte del CLSI para la detección de la RAN a gentamicina en Enterococcus spp. Según los criterios de EUCAST, la detección de estas cepas se puede realizar utilizando un disco de gentamicina con una carga de 30μg y si el halo de inhibición es <8mm, se considera que la cepa presenta RAN a gentamicina (CMI >128mg/L)2.
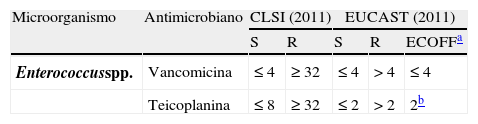
Resistencia a los glucopéptidosSe han descrito 2 tipos de resistencia a los glucopéptidos en enterococo: 1) resistencia intrínseca: en las especies Enterococcus gallinarum (gen vanC1), Enterococcus casseliflavus (gen vanC2) y Enterococcus flavescens (gen vanC3). Se caracteriza por conferir bajos niveles de resistencia a la vancomicina (CMI 2-32mg/L) según los criterios del CLSI (tabla 3)1. Asimismo estas cepas son sensibles a la teicoplanina (CMI 0,5-2mg/L). Los genes se localizan en el cromosoma bacteriano y no son transferibles por lo que tienen escasa trascendencia clínica y epidemiológica; 2) resistencia adquirida: se han descrito 8 fenotipos diferentes (VanA, VanB, VanD, VanE, VanG, VanL, VanM y VanN) mediados por los operones de genes de resistencia vanA, vanB, vanD, vanE, vanG, vanL, vanM y vanN, respectivamente. Los fenotipos VanA y VanB son los más frecuentes, mientras que VanD, VanE, VanG, VanL, VanM y VanN son infrecuentes y no se han detectado hasta la fecha en España17,25–28.
El fenotipo VanA se caracteriza por presentar elevados niveles de resistencia a la vancomicina y a la teicoplanina (CMI 16-1.024mg/L), es de carácter inducible (por vancomicina y teicoplanina) y es el más frecuente entre los aislamientos clínicos de enterococos resistentes a la vancomicina. El fenotipo VanB se caracteriza por presentar moderados niveles de resistencia a la vancomicina, CMI 4-32mg/L y sensibilidad a la teicoplanina (CMI 0,5-1mg/L) aunque se han descrito cepas con altos niveles de resistencia a la vancomicina (CMI 1.024mg/L). El fenotipo VanB es inducible por vancomicina y la presencia de vanB2 en particular se describe cada vez con más frecuencia entre los aislados resistentes a este compuesto25.
Detección fenotípica de la resistencia a los glucopéptidosLa detección de los fenotipos VanA y VanM en el laboratorio es sencilla, sin embargo, existen algunos problemas para la detección de los otros fenotipos (VanB a VanN) por los bajos niveles de resistencia a la vancomicina que pueden conferir (a veces en la categoría de sensible). En estos casos es importante la detección del mecanismo de resistencia mediante técnicas moleculares como la PCR con cebadores específicos. Los métodos fenotípicos que se emplean para detectar la resistencia a glucopéptidos en enterococos incluyen: 1) difusión con discos en medio agar Mueller Hinton. Este método tiene bajo poder discriminatorio, sobre todo con los valores intermedios de CMI, 8-16mg/L y su realización debe evitarse siempre que sea posible. La lectura del antibiograma se debe hacer a las 24h completas de incubación y con luz transmitida, considerando como resistente la presencia de cualquier crecimiento o velo dentro del halo de inhibición. En caso de duda debe realizarse la determinación de las CMI de vancomicina y de teicoplanina mediante métodos de dilución o mediante Etest6; 2) microdilución y dilución en agar. Para ello se utiliza el medio Mueller Hinton con concentración ajustada de cationes (Ca+2 y Mg+2), un inóculo estándar (0,5 de la escala McFarland) e incubación de 24h a 35±2°C6; 3) método de cribado: el CLSI recomienda el uso de una placa de agar BHI con 6mg/L de vancomicina, un inóculo de 105-106UFC/depósito (1-10μl de un inóculo equivalente al 0,5 escala McFarland) y una incubación de 24h a 35±2°C. La detección de más de 1 colonia, de un ligero o de un claro crecimiento son indicativos de resistencia a la vancomicina1, y 4) empleo de medios de agar cromogénico comercializados para el aislamiento selectivo de cepas de enterococo resistentes a la vancomicina que permiten realizar estudios de vigilancia epidemiológica6,17.
En la tabla 5 se indican los puntos de corte definidos por el CLSI y el EUCAST para la detección de resistencia a glucopéptidos en enterococos.
Resistencia a los antimicrobianos en Streptococcus pneumoniaeResistencia a betalactámicosLa resistencia de S. pneumoniae a la penicilina y la sensibilidad disminuida a otros betalactámicos como las cefalosporinas de tercera generación se deben a cambios estructurales en las dianas de acción de estos compuestos, es decir, en las penicillin binding proteins (PBP, ‘proteínas fijadoras de penicilina’). Las principales PBP implicadas en la resistencia a los betalactámicos en este microorganismo son las PBP 1a, 2b y 2x. En estos casos, los genes que las codifican tienen una estructura en mosaico como resultado de que una parte de ellos ha sido reemplazada por variantes alélicas de genes de otras cepas de S. pneumoniae o de estreptococos del grupo viridans. Las alteraciones en las PBPs 2b y 2x confieren resistencia de bajo nivel a penicilina. Los cambios en la PBP 1a confieren resistencia de alto nivel a la penicilina en aquellos aislamientos que tienen alterada la PBP 2x o las dos (PBP 2b y PBP 2x). Las cefalosporinas de tercera generación no se unen a la PBP 2b por lo que la resistencia a estos compuestos se debe a cambios en las PBPs 2x y 1a. Los neumococos resistentes a la penicilina presentan menor afinidad por los otros antimicrobianos betalactámicos29.
Detección fenotípica de la resistencia a los betalactámicosLa utilización de los discos de penicilina (10 U), cefotaxima, ceftriaxona y cefepima está desaconsejada para determinar la sensibilidad de S. pneumoniae frente a betalactámicos. El cribado mediante difusión con discos de oxacilina (1μg) se considera el mejor método para detectar aquellas cepas con CMI de penicilina superiores a 0,06mg/L. La sensibilidad a la oxacilina implica sensibilidad a todos los betalactámicos1,2. Para su determinación se debe realizar una suspensión bacteriana en medio líquido estéril a partir de 3-4 colonias aisladas, ajustar el inóculo a una turbidez equivalente al 0,5 de la escala McFarland, inocular una placa de Mueller Hinton con sangre (MH-S) y colocar el disco mencionado. Tras una incubación durante 18-24 horas en la estufa de CO2 a una temperatura de 35±2°C (o en jarras de incubación con atmósfera de 5% de CO2 e incubación en estufa convencional) se procede a la lectura del halo de inhibición.
Por su parte, la CMI por microdilución es el método de referencia y requiere el empleo de caldo de Mueller Hinton con concentración ajustada de cationes (Ca+2 y Mg+2) y 5% de sangre lisada de caballo, un inóculo estándar (0,5 de la escala McFarland) e incubación de 20-24h a 35±2°C en atmósfera normal1,2,6.
Valoración y expresión de resultados de la difusión con disco de oxacilinaHalo de inhibición de oxacilina≥20mm: cepa sensible a la penicilina y a la cefotaxima/ceftriaxona. Resultado definitivo.
Halo de inhibición de oxacilina<20mm: cepa con posibles alteraciones en las PBP. Se recomienda determinar la CMI a la penicilina y a la cefotaxima mediante Etest o microdilución1,2.
Valoración y expresión de resultados de la CMI1,2,30PenicilinaAislamientos meníngeos:
- •
CMI de penicilina≤0,06mg/L: informar como sensible a la penicilina.
- •
CMI de penicilina≥0,12mg/L: informar como resistente a la penicilina.
Aislamientos no meníngeos:
- •
CMI de penicilina≤0,06mg/L. Informar como sensible a la penicilina. Estas cepas también son sensibles a la ampicilina por vía oral o parenteral y a todos los betalactámicos.
- •
CMI de penicilina≥0,12mg/L y≤2mg/L. Informar como sensible a la penicilina (parenteral). Estas cepas también son sensibles a amoxicilina, amoxicilina/ácido clavulánico, cefotaxima, ceftriaxona, cefepima y ertapenem.
Insertar una recomendación terapéutica: «cepa con CMI de penicilina≤2mg/L. En infecciones no meníngeas, pueden utilizarse como tratamiento las penicilinas endovenosas a altas dosis. No utilizar penicilina en caso de meningitis».
En caso de que se vaya a utilizar penicilina por vía oral informar: a) CMI de penicilina≤0,06mg/L: sensible; b) CMI de penicilina 0,12-1mg/L: intermedio; c) CMI de penicilina≥2mg/L: resistente.
- •
CMI de penicilina=4mg/L. Informar como «resistencia intermedia» a la penicilina (parenteral). Insertar una recomendación terapéutica: «en caso de infección meníngea, el aislamiento se considera resistente a la penicilina».
- •
CMI de penicilina>4mg/L. Informar como resistente a la penicilina (parenteral).
En los aislamientos no meníngeos se deben informar tanto las interpretaciones para meningitis como las de no meningitis.
Aislamientos meníngeos:
- •
CMI de cefotaxima≤0,5mg/L. Informar como sensible a la cefotaxima.
- •
CMI de cefotaxima=1mg/L. Informar como resistencia intermedia a la cefotaxima.
- •
CMI de cefotaxima≥2mg/L. Informar como resistente a la cefotaxima.
Aislamientos no meníngeos:
- •
CMI de cefotaxima≤1mg/L. Informar como sensible a la cefotaxima. En los aislamientos con CMI de 1mg/L insertar una recomendación terapéutica: «en caso de infección meníngea el aislamiento se considera con resistencia intermedia a la cefotaxima».
- •
CMI de cefotaxima=2mg/L. Informar como resistencia intermedia a la cefotaxima. Insertar una recomendación terapéutica: «en caso de infección meníngea el aislamiento se considera resistente a la cefotaxima».
- •
CMI de cefotaxima≥4mg/L. Informar como resistente a la cefotaxima.
En los aislamientos no meníngeos se deben informar tanto las interpretaciones para meningitis como las de no meningitis.
Resistencia a las fluoroquinolonasEl principal mecanismo de resistencia a las fluoroquinolonas en S. pneumoniae es debido a mutaciones en las regiones determinantes de resistencia a quinolonas (QRDR) tanto de la ADN topoisomerasa IV (ParC y ParE) que es la diana primaria, como de la ADN girasa (GyrA y GyrB) que es la diana secundaria. La resistencia también puede adquirirse por recombinación interespecies con otros estreptococos, sin embargo, este mecanismo es poco frecuente31.
Detección fenotípica de la resistencia a las fluoroquinolonasLa detección de las cepas que presentan mutaciones de primer nivel es importante para evitar fallos en el tratamiento antimicrobiano. El Comité de l’Antibiogramme de la Société Française de Microbiologie (SFM, 2010) recomienda la utilización de discos de norfloxacino (5μg) como método de cribado para detectar cepas de neumococo con mutaciones de primer nivel, utilizando como punto de corte de resistencia a aquellos halos de<10mm (tabla 6)3. El medio utilizado es el agar Mueller-Hinton con 5% de sangre de carnero y se requiere una incubación de 20-24h en atmósfera enriquecida con 5% de CO2. El principal inconveniente es la posibilidad de obtener falsa resistencia (halos inferiores a 10mm) en aquellas cepas con fenotipo mucoso como las del serotipo 3. Esta técnica puede realizarse directamente a partir de las botellas de hemocultivo con crecimiento positivo en las que se sospeche la presencia de S. pneumoniae6.
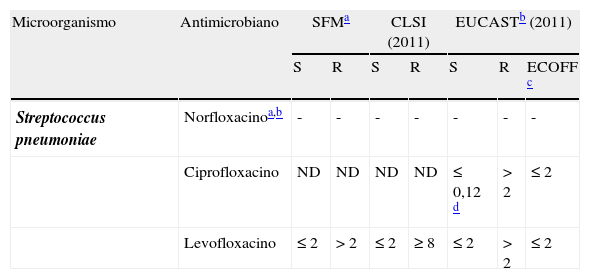
Puntos de corte (CMI, mg/L) para ciprofloxacino y levofloxacino frente a Streptococcus pneumoniae1–3
| Microorganismo | Antimicrobiano | SFMa | CLSI (2011) | EUCASTb (2011) | ||||
| S | R | S | R | S | R | ECOFF c | ||
| Streptococcus pneumoniae | Norfloxacinoa,b | - | - | - | - | - | - | - |
| Ciprofloxacino | ND | ND | ND | ND | ≤0,12 d | >2 | ≤2 | |
| Levofloxacino | ≤2 | >2 | ≤2 | ≥8 | ≤2 | >2 | ≤2 | |
Disco de 5μg, solo como cribado: halo<10mm) y/o CMI de norfloxacino >16mg/L indican riesgo elevado de selección in vivo de mutantes resistentes a fluoroquinolonas y de fracaso clínico.
El último documento de EUCAST indica que las cepas salvajes de S. pneumoniae no se deben considerar sensibles a ciprofloxacino y por lo tanto se deben categorizar como de «sensibilidad intermedia» a esta fluoroquinolona2. Por su parte, el CLSI nunca ha incluido a ciprofloxacino entre los antimicrobianos cuya sensibilidad debe estudiarse en el antibiograma de S. pneumoniae (tabla 6)1. Sin embargo, el valor de CMI de ciprofloxacino de 4mg/L propuesto por Chen et al. es el más utilizado para detectar cepas con mutaciones de primer nivel cuando se realiza microdilución en caldo (la técnica empleada es la misma que la descrita para betalactámicos)32.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.