La lepra es una infección granulomatosa crónica de distribución mundial1 causada por Mycobacterium leprae y M. lepromatosis2. Se puede clasificar, según el número de lesiones cutáneas, en dos grupos3: a) grupo paucibacilar (con menos de 6 lesiones, sin presencia de BAAR en biopsias), que incluye las formas clínicas indeterminada, tuberculoide tuberculoide (TT) y borderline tuberculoide (BT), y b) grupo multibacilar (con 6 o más lesiones, y presencia de BAAR en la biopsia), en el que se agrupan las formas borderline borderline (BB), borderline lepromatosa (BL), la lepra lepromatosa (LL) y la lepra histiode, siendo esta última una forma muy poco frecuente que puede presentarse como reactivación por tratamiento incompleto o más raramente como forma inicial de esta enfermedad4.
Dado que, M. leprae no es cultivable in vitro, el actual desarrollo de técnicas moleculares, entre ellas la secuenciación de los genes 16S rARN o hsp655-7, son de especial utilidad para el diagnóstico de confirmación de esta enfermedad en presentaciones atípicas.
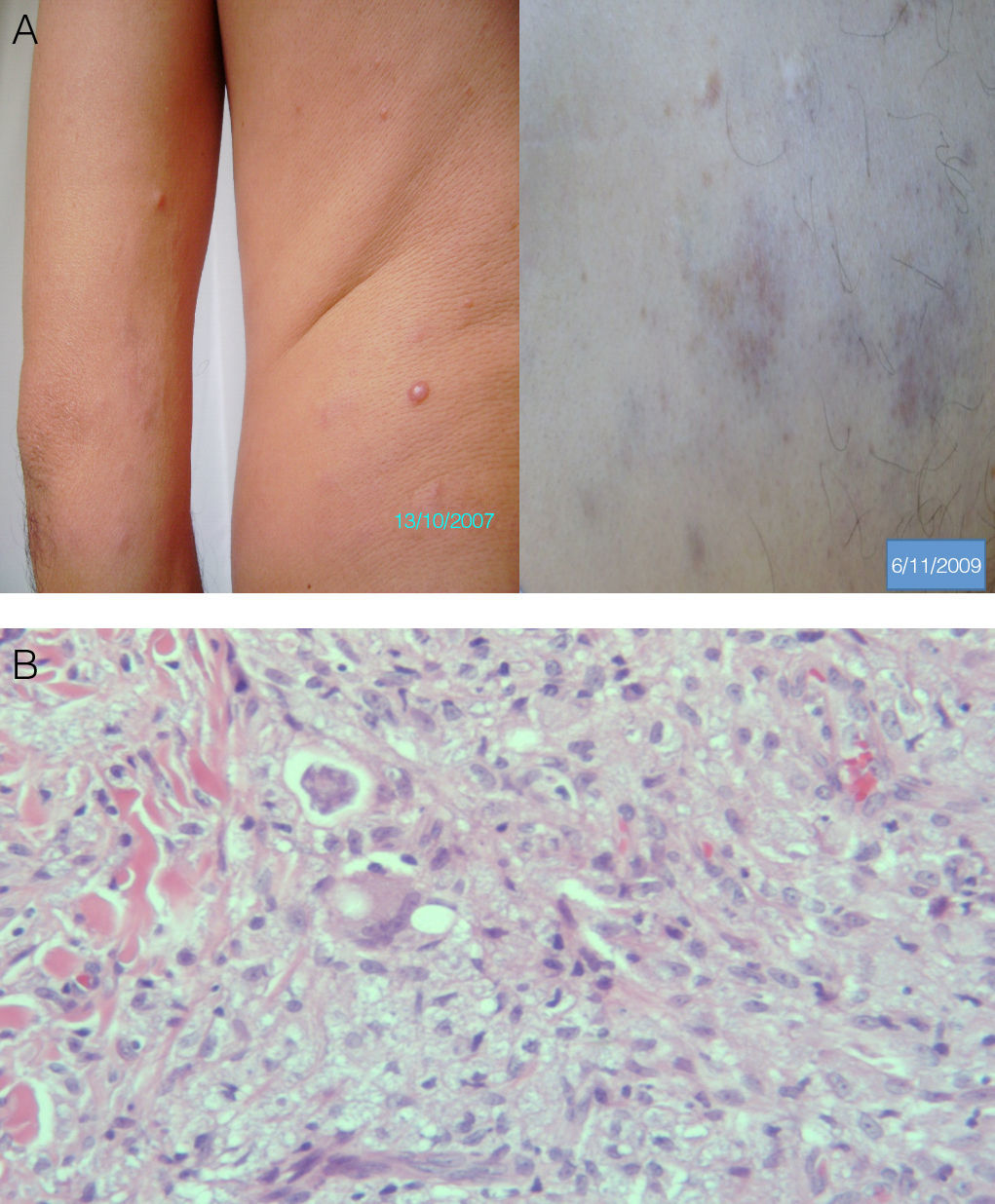
En esta carta comunicamos cómo se realizó el diagnóstico tardío de un caso inicial de lepra histioide en un inmigrante de origen paraguayo. Se trataba de un varón de 42 años de San Lorenzo (Paraguay) que, tras un semestre de residencia en Almería (España), le aparecieron nódulos lenticulares, indoloros, diseminados por el tronco y las extremidades (fig. 1A) y que motivaron la consulta a su médico de atención primaria, quien, ante la mala evolución clínica tras 8 meses de terapia esteroidea —extensión de las lesiones y aparición de placas eritematovioláceas disestésicas (dolorosas a la presión) en muslos y tobillos—, decidió remitirlo a la consulta de dermatología.
En la consulta especializada se plantearon inicialmente los posibles diagnósticos de granuloma perforante diseminado, sarcoidosis, eritema nodoso, tuberculosis-micobacteriosis cutánea y sífilis secundaria, solicitándose las pruebas complementarias pertinentes: radiografía de tórax, perfil bioquímico-hormonal, ANA, serología de lúes, biopsia de lesiones y cultivo bacteriano y estudio de micobacterias.
Los hallazgos histológicos —presencia de un infiltrado superficial histiocitario globoide sin granulomas y zonas de necrosis focal con intenso infiltrado linfocitario y abundantes microorganismos (Ziehl-Nielsen negativos y de Fite-Faraco positivos) (fig. 1B)— y microbiológicos—presencia de abundantes BAAR uniformemente teñidos, más largos que el morfotipo habitual del bacilo de Hansen, ausencia de globis y cultivo de micobacterias negativo— enfocaron el diagnóstico hacia una más probable micobacteriosis cutánea diseminada, no descartándose una posible forma atípica de lepra.
Para resolver la duda, se planteó la identificación molecular de los bacilos no cultivables presentes en la biopsia cutánea mediante PCR universal del gen 16S rARN (amplificación de 1.500 pb). Para ello, se secuenciaron las primeras 500 pb del extremo 5′ y la secuencia obtenida se alineó en GenBank mediante software BIBI (http://umr5558-sud-str1.univ-lyon1.fr/lebibi/lebibi.cgi), encontrando una similitud del 99,6% entre las secuencias de las cepas problema y la secuencia FM211192 de M. leprae. Además, se confirmó este resultado por amplificación y secuenciación de 440 pb del gen hsp657,8, obteniendo una similitud de alineamiento del 100% del fragmento problema con las secuencias FM211192, AY299192 de M. leprae depositadas en Genebank. La revisión del cuadro clínico, junto con esta identificación molecular, permitió la confirmación diagnóstica de lepra histioide, iniciándose tratamiento específico con MDT-COMBI® (rifampicina, clofamicina y dapsona).
Durante el seguimiento se realizaron varios cambios terapéuticos; el primero a los 10 meses, sustituyendo la clofamicina por ofloxacino en la terapia combinada por sospecha de resistencia antimicrobiana, dada la persistencia de abundantes BAAR en la biopsia; y un segundo, transcurrido un año, con reintroducción de la pauta inicial al comprobar molecularmente, mediante una técnica de hibridación reversa (GenoType®LepraeDR. Hain Lifescience GmbH Nehren, Alemania) que los BAAR presentes en una nueva biopsia presentaban una mutación en el gen gyrA asociada a resistencia al ofloxacino9,10. No podemos dar información sobre el resultado final del tratamiento debido a que no acudió a las revisiones periódicas a partir de marzo del 2010.
Los motivos por los que el diagnóstico se retrasó se justifican por su manifestación inicial en el país de destino, donde la enfermedad es excepcional, confundiéndose inicialmente con una dermatitis inespecífica y después con una posible micobacteriosis cutánea; dadas su presentación atípica y la observación microscópica de BAAR diferentes al morfotipo habitual de M. leprae. La abundante carga bacilar de la biopsia permitió la identificación molecular de M. leprae y el diagnóstico de lepra en su variedad histioide.
FinanciaciónEste estudio ha sido parcialmente financiado por la Junta de Andalucía (PI: 0444/2008 y 0306-2009) y gracias al FIS (IF01-3624, IF08-36173).
A los Servicios de Análisis Clínicos (Microbiología), Anatomía Patológica y Dermatología del Hospital «La Inmaculada», Huércal-Overa, Almería; en especial a las Dras. Lidia Olaz-Cecilia, Pilar Luzón García y Mercedes Llamas-Pérez, quienes aportaron la información clínica-anatomopatológica y el material biológico y fotográfico. También a la Dra. Mercedes Morales Torres por su capacidad de síntesis en la exposición de datos preliminares en la reunión SEIMC 2010.
Parte de este trabajo ha sido aceptado como póster en el Congreso SEIMC celebrado en Barcelona del 19 al 22 de mayo de 2010.